Bases Químicas de la Herencia: ADN, ARN y Dogma Central de la Universidad Peruana Cayetano Heredia
Diapositivas de la Universidad Peruana Cayetano Heredia sobre Bases Químicas de la Herencia: ADN, ARN y Dogma Central. El Pdf explora las estructuras de nucleótidos, fosfatos, pentosas y bases purínicas/pirimidínicas, con nomenclatura de ácidos nucleicos. Es un recurso de Biología para estudiantes universitarios.
Ver más45 páginas

Visualiza gratis el PDF completo
Regístrate para acceder al documento completo y transformarlo con la IA.
Vista previa
Ácidos Nucleicos
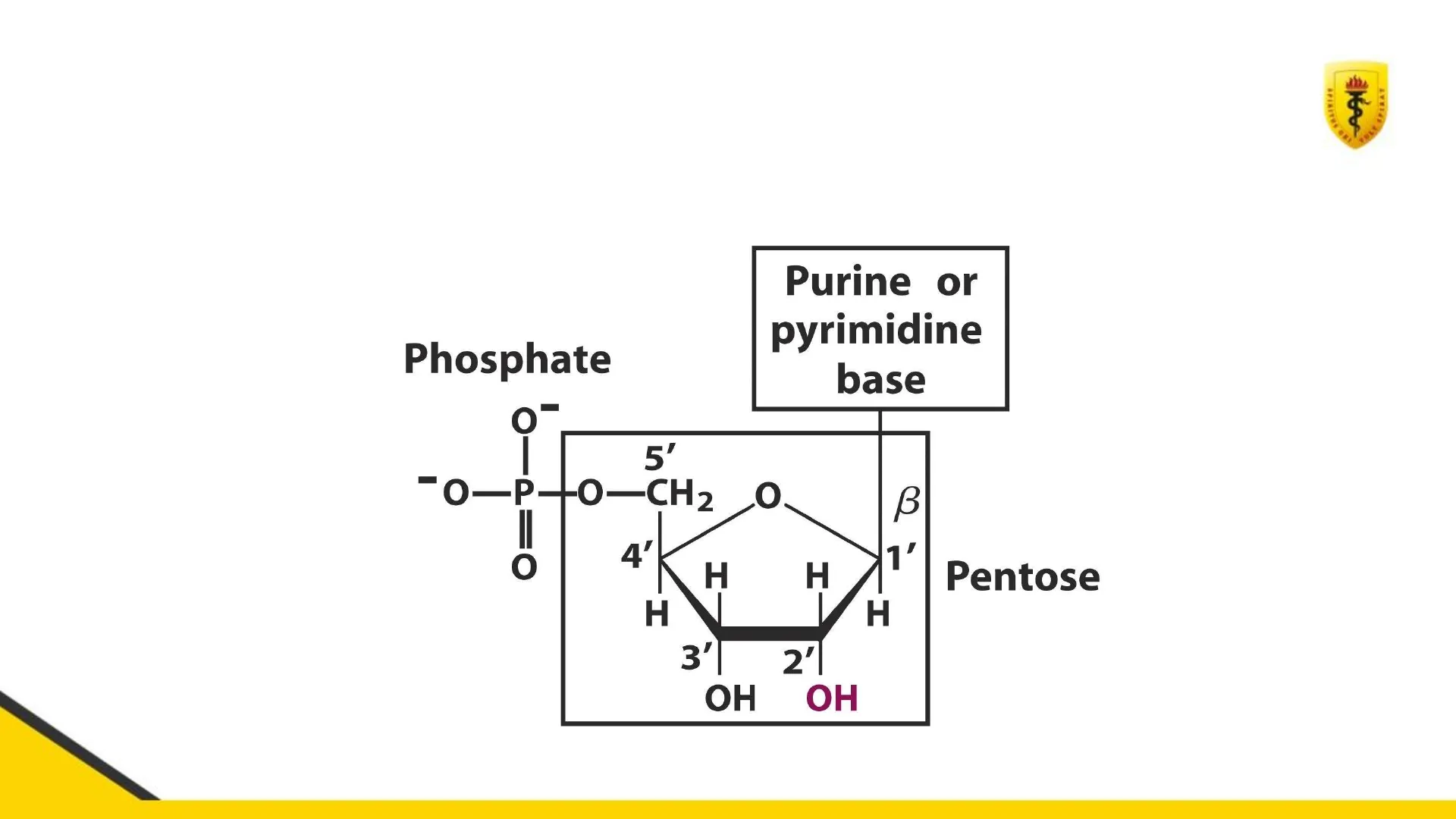
SPIRITUS UBI VULT SPIRAT UNIVERSIDAD PERUANA CAYETANO HEREDIA Bases químicas de la herencia: ADN, ARN y dogma central Dr. Jorge Rodríguez Bailón Fundamentos de Genética para Ciencias Veterinarias 2025 Facultad de Medicina Veterinaria y ZootecniaACIDOS NUCLEICOS THAY SPIRAT Phosphate Purine or pyrimidine base I 5' -O-P-+O-CH2 .O B 4' 1' O H H Pentose H H 3' 2'| OH OH
Estructura Primaria del DNA vs. RNA
Es la secuencia de nucleotidos unidos por enlaces covalentes de tipo fosfodiester DNA 5' End O 0-P=0 0-P=0 O U 5' CH ___ O H H H H 3' 0 OH 0-P=0 Ò G 5' CH2 0 5' CHO H H H H H H 3' OH 0-P=0 0-P=0 O-A-O- C 5' 5' CH2_O H H H H 3' O OH 0-P=0 - 5' in >00 3' 5'CH ___ O H H H H 3 O H 0-P=0 RNA 5' End O O A I 5' CH2 O H H H H 3 O H Enlace fosfodiester O-A-O 0-P=0 T H H 3 H 0-A-O- G 10700 3' THAY SPIRATAzucar THAY SPIRAT OH OH 5' CH2 OH OH - 4' 4' 3' 3' 2 OH OH OH Ribose 2'-Deoxyribose Figure 2.2 Nucleic acid sugars 5' CH2
Numeración de Carbonos de la Pentosa
THAY SPIRAT Phosphate Purine or pyrimidine base 5' -O-P-+O-CH2 O B 4' 1' 0 H H Pentose H H 3' 2' OH OH Ribosa - 2 grupos OH (en C2' y en C3') Desoxi rribosa (no tiene OH en C2') Pueden tener 1, 2 ó 3 grupos fosfato (mono-, di- o tri- fosfato)Fosfatos: Y β 1 NH2 N- C N 0- HC 1 C CH N N = Ö = Ö Ö I H H OH OH Adeninosina trifosfato (ATP) THAY SPIRAT 1 "O-P-O-P-O-P-O-CH2 O -TABLE 8-1 Nucleotide and Nucleic Acid Nomenclature Base Nucleoside Nucleotide Nucleic acid DEAF SPIRAT Purines Adenine Adenosine Adenylate RNA Deoxyadenosine Deoxyadenylate DNA Guanine Guanosine Guanylate RNA Deoxyguanosine Deoxyguanylate DNA Pyrimidines Cytosine Cytidine Cytidylate RNA Deoxycytidine Deoxycytidylate DNA Thymine Thymidine or deoxythymidine Thymidylate or deoxythymidylate DNA Uracil Uridine Uridylate RNA Nomenclatura abreviada : Ribonucleótidos trifosfato: ATP, CTP, GTP, UTP Desoxirribonucleicos trifosfato: dATP, dCTP, dGTP, dTTP
Bases Nitrogenadas en Ácidos Nucleicos
THAY SPIRAT NH2 0 C C N N C HN C II CH CH HC C C C N N H2N N H Adenine Guanine Purines NH2 0 0 O= C C C CH3 N CH HN HN CH II C CH C CH C CH 0 N O N O N H H H Cytosine Thymine (DNA) Uracil (RNA) Pyrimidines N H NEl esqueleto del DNA consiste en la unión covalente de un azúcar pentosa y grupos fosfatos alternados base1 base2 basez O O O 0 O O 0 0 O `P P P O O 0 O O O Sugar Phosphate - THAY SPIRAT
Secuencias de Bases: Dirección 5' -> 3'
LAS SECUENCIAS DE BASES SE ESCRIBEN DE 5' -> 3' THAY SPIRAT A C G T A 5' End 3' End P P P P P OH IMPORTANTE: v La síntesis de ácidos nucleicos (DNA y RNA) ocurre de 5' a 3' v Durante la traducción, la lectura de la secuencia de bases del RNA ocurre de 5'a 3'
Estructura Secundaria del DNA
Dos cadenas antiparalelas forman una doble hélice de giro hacia la derecha, girando una vuelta completa cada 10 pares de bases. Ţ 3.4 Å Minor groove F 36 Å Major groove 20 Å (b) (c) (a)MODELO DE DNA WATSON & CRICK 1953) 1. Dos cadenas antiparalelas 2. Fosfatos hacia fuera 3. Bases hacia adentro 4. Bases complementarias: Purina - Pirimidina Adenina - Timina Guanina - Citosina 5. Fuerzas estabilizadoras: Puentes de hidrógeno -específica Fuerzas hidrofóbicas Apilamiento de bases inespecíficas 6. Una vuelta completa cada 10 bases 5' 3' C G A T A T T A C G G C T A C G A T 3' 5'5' 3' C G A T N O 1 C N- NIN H-N C C-1' 1 Ö N-C C N= H C-1' -11.1 Å H H 1 1 C-C H ₭-3.0 Å-> Cytosine N C - C- H-C 11 C-1' 1 -C N Nº / C-1' ₭-2.9 Å-> H -10.8 Å- A T 3' 5' Adenine T A C G G C N-H I N -N C Guanine 1 T A C G CH3 <2.8Å-> O H H WI C C H -3.0 Å- N 11 H-C A T II Thymine THAY SPIRAT k-2.9 Å-> OIN H-N =C
RNA
Propiedades del RNA
RNA THAY SPIRAT Propiedades: O -CH2 Base 4 1' H H H H 3' 2' OH OH Ribose · Tiene un ribosa · Hebra simple · Formados por las bases: Adenina, Guanina, Citosina y Uracilo (en vez de Timina) · Catalizador Biológico àRibozima
Tipos de RNA
RNA THAY SPIRAT TIPOS • mRNA es el que influencia el fenotipo Expresión genica Intermediario DNA - ProteínaRNA THAY SPIRAT TIPOS · RNA funcional Procarlotas y Eucarlotas Paso de la información Procesamiento otros RNA Regulación niveles de RNA y Prot tRNA rRNA Específicos de Eucariotas Procesamiento del RNA snRNA miRNA SİRNA Específicos de Eucariotas Supresión de la expresión de genes Mantener la estabilidad del genoma
Estructura del RNA
THAY SPIRAT (a) Secondary structure Hairpin ( Double-helical stem region Stem-loop (b) Tertiary structure 3' 3' Loop 1 Stem 2 Stem 1 Loop 2 5' 5' PseudoknotC G A C G A-160 `G C. C C_ `G GC G GU-A A-1 G-C C-G A 140 G G . U. 0-GA AC-GGU 180 A A A A G A G A CCACGCCA G 200 100 CGCGG CUGG 120-G C CA G A A C- C 220 U A G A OGG G A 0 G- 0 AG CG-CCUC U-A G-C-240 G-C GOAG- G G AUAC- G AUA 260 GT GGGUUCA GUACGG C 80 .A A -G 00 C G C G GALG U G-300 C U U GA U AGCCAGUGAG O 1 C C-G A G U-A G- _C A-U C- G N N N Uracil N= NH2 U A GGCC .CA Algunos RNAs también presentar estructuras secundarias THAY SPIRAT G GU GGUGCG G C C A 1 G GUGCC GAC ,A ACCC -G C- A 280 CCCAAGUCAUGCC COGGG-CAAGG 000000 A A A 60 A 20 CG . UAAGUAG A CCGGUCGUU C GGGGGA GACG GOGGAG GG UCUCCU CUGC UGCUUCG A-U 1 GAAGCUGACCAG-CC A CUUUGACUGG C A A C C G A C A U-377 360-U G Guanine Los RNA ribosomales presentan múltiples regiones que forman estructuras secundarias. Puede darse la formación de pares G-U en los RNAS 40 SC 1 `330 OUIHIN C AA I G G GRNA de transferencia (tRNA) Acceptor stem 5' end 3' end THAY SPIRAT TỤC arm Amino acid arm 64 1 5' 54 56 72 3' D arm (residues 10-25) 20 12 44 26 38 Anticodon arm Anticodon 32 (a) Anticodon stem
Dogma Central de la Biología Molecular
THAY SPIRAT Procesos esenciales: Dogma central de la BM replication (DNA -> DNA) RNA protein transcription (DNA -> RNA) RNA Polymerase RNA Transport to cytoplasm for protein synthesis (translation) mRNA translation (RNA -> Protein) Ribosome Protein DNA mRNA Transcription Maze MRNA -- DNA DNA Polymerase DNA
Replicación del ADN
THAY SPIRAT · Fiel y rápida · 1 error/millon bp produce 6400 errors cada vez que se divide una célula. · E. coli replica su DNA a un ratio de 1000 nucleotidos/seg.THAY SPIRAT (a) Conservative replication (b) Dispersive replication (c) Semiconservative replication Original DNA First replication V Second replication Figure 12-1 Genetics: A Conceptual Approach, Third Edition 2009 W.H. Freeman and Company
Experimento de Meselson & Stahl
Meselson & Stahl THAY SPIRAT · Dos isotopos de nitrógeno: · 14N forma común; 15N forma rara pesada . E. coli crecio en un medio con 15N y luego fue transferida a un medio con 14N · Cultivos de E. coli fueron separados por una gradiente de centrifugaciónExperiment Question: Which model of DNA replication-conservative, dispersive, or semiconservative- applies to E. coli ? (a) (b) (c) (d) Method Transfer to 14N medium and replicate Replication in 14N medium Replication in 14N medium Spin Spin Spin Spin Results Light (14N) Heavy (15N) DNA from bacteria that had been grown on medium containing 15N appeared as a single band. After one round of replication, the DNA appeared as a single band at intermediate weight. After a second round of replication, DNA appeared as two bands, one light and the other intermediate in weight. Samples taken after additional rounds of replication appeared as two bands, as in part c. Original DNA Parental New strand strand Conclusion: DNA replication in E.coli is semiconservative. Figure 12-3 Genetics: A Conceptual Approach, Third Edition 2009 W. H. Freeman and Company THAY SPIRAT 15N medium
Etapas de la Replicación Procariota
Etapas de la replicación procariota THAY SPIRAT · Inicio de replicación: 245 bp- oriC • Elongación: DNA polimerasa III • Remoción del primer RNA: DNA polimerasa I • Union de los extremos del DNA: DNA ligasa
Función de las Enzimas en la Replicación
Enzyme Function DEAF SPIRAT DNA Gyrase (Topoisomerase) Stabilizes DNA helix as it is unwound by Helicase Single Stranded Binding Proteins Hold unzipped, single-stranded sections of DNA apart during replication Helicase Unwinds DNA helix and unzips strands by breaking Hydrogen bonds DNA Polymerase III Adds new DNA nucleotides in the 5' > 3' direction DNA Primase Adds primers of RNA nucleotides to the lagging strand as starting points for replication DNA Polymerase I Replaces RNA primers with DNA nucleotides DNA Ligase Joins Okazaki fragments on lagging standLVELAS ATRA DNA primase RNA primer DNA ligase DNA Polymerase (Pola) 3' Lagging strand 3' 5' Okazaki fragment 5' 5' Leading strand Topoisomerase 3' DNA Polymerase (Pol8) Helicase Single strand, Binding proteins
Replicación Eucariota Lineal
Linear eukaryotic replication THAY SPIRAT Origin 5' 3' 3' 5' Leading strand Lagging strand Lagging strand 3' 5' 3' 5' Leading strand Unwinding and replication Origin Unwinding and replication Figure 12-10c Genetics: A Conceptual Approach, Third Edition @ 2009 W. H. Freeman and Company · Miles de sitios de origen de replicación, un replicon mide entre 200,000 ~ 300,000 bp
Características de la Replicación del DNA Eucariota
Características de la replicación del DNA eucariota THAY SPIRAT · Requiere de un segmento de doble hebra de DNA molde, enzimas y nucleótido · La dirección de la replicación es 5'-> 3'. · La DNA polimerasa añade nucleótidos solo en el extremo 3 de la hebra en crecimiento
Transcripción y Traducción
THAY SPIRAT Trascripción y Traducción
Flujo de Información Genética
Flujo de Información Genética Dogma central de la Blología Molecular THAY SPIRAT DNA mRNA Polypeptide 5' 3' 5' Amino terminus Replication THIA U J GHIC G GINIC G Gly Transcription AIIT A THIA U AIIT A Tyr CHIG C RNA AIIT A CHIG C Thr THIA U TILIA U TIIA U Phe TILIA U GILIC G CHIG c Ala CIIG GHIC G TIA U Val THIA THIA U CHIG C Ser THIA 3' Î Ï 5' 3' Carboxyl terminus Template strand DNA replication V DNA Translation Transcription Reverse transcription Protein RNA RNA replication Translation Protein C G Arg
Información Genética
THAY SPIRAT INFORMACIÓN · Toda información contenida carece de utilidad si no se mantiene en orden y sin errores.
Principios de la Transcripción
Principios de la Transcripción · Complementariedad de Bases (A-T)(G-C) CH3 k-2.8 Å->| 1 H C -H N C 1 Thymine N C NINH-N N 11 C H-C C-1' 1 Ö N N H C-1' H H ₭-2.9 Å-> 1 -N O IN H C- H -3.0 Å- N N C N-H III N C H-C C-1' 1 C C N N C-1' -2.9 Å->| H -10.8 Å- · Proteínas de unión a ácidos nucleicos Adenine 1 -C 11.1 Å- C Cytosine -- Guanine II N-HILO THAY SPIRAT C k-3.0 Å->
Interacciones DNA y Proteína
Las interacciones entre DNA y Proteína usualmente involucra cierto grado de especificidad de secuencia THAY SPIRAT (b) Bacteriophage 434 repressor Q S Q Q T Muchas proteínas que interactúan con el DNA en las hendiduras mayores del DNA