Biologia Molecolare: Tecniche di Analisi per l'Espressione Genica
Slide dall'Università sulla Biologia Molecolare Tecniche di Analisi. Il Pdf esplora diverse tecniche di analisi in biologia molecolare, come la denaturazione del DNA e i microarray, con schemi e illustrazioni utili per lo studio universitario di Biologia.
Mostra di più59 pagine

Visualizza gratis il Pdf completo
Registrati per accedere all’intero documento e trasformarlo con l’AI.
Anteprima
Tecniche di Analisi
Spettro di Assorbimento del DNA
il dsDNA possiede legami non covalenti deboli (legami idrogeno tra basi complementari e interazioni idrofobiche dovute all'impilamento delle basi. I legami sono indeboliti da vari agenti: · alta temperatura · alto pH · agenti chimici
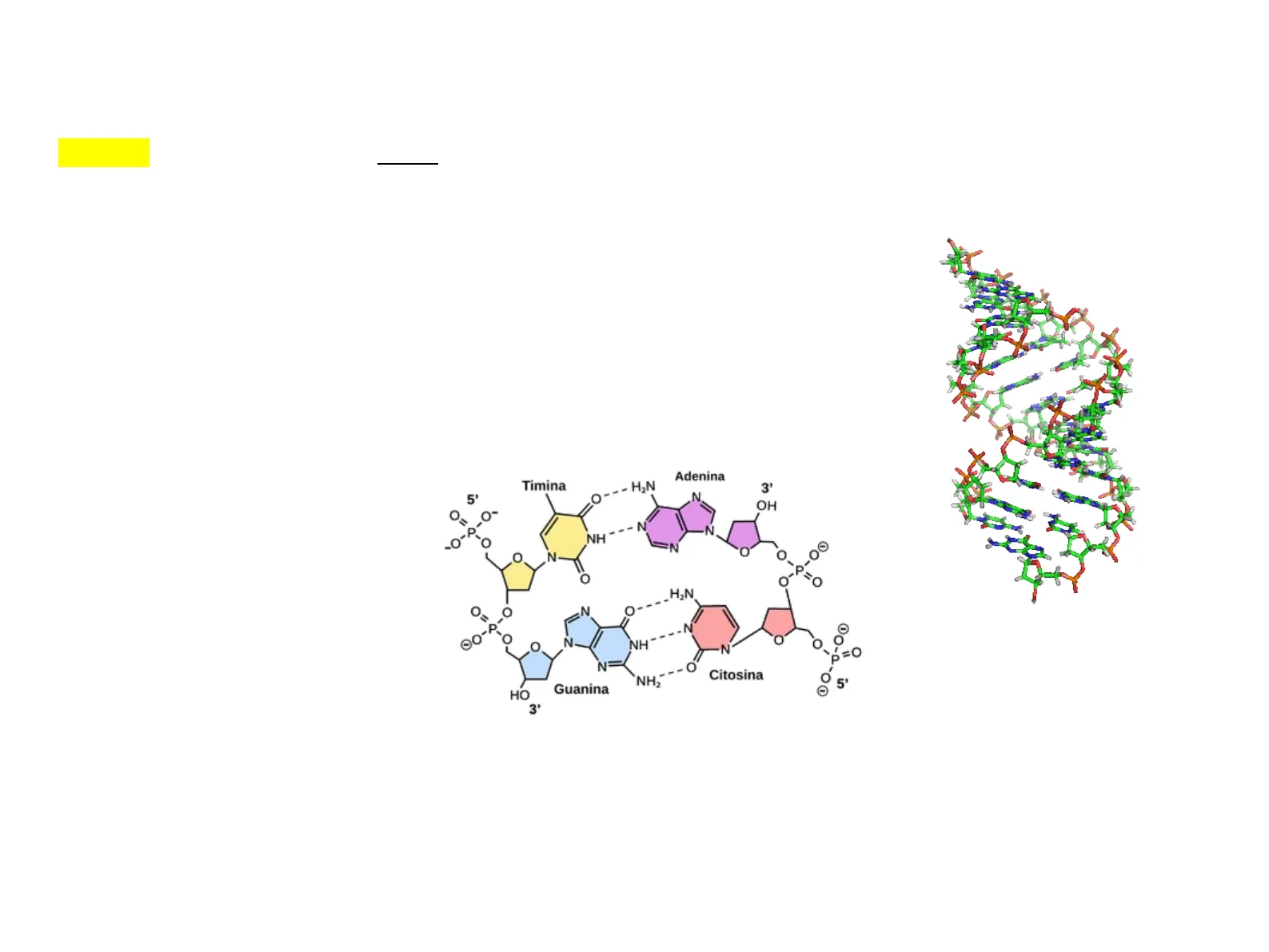
Adenina Timina -- - H2N N OH -- N O C EN O 0 N O H,N Os 0 N 0 0- 00- N NH N - =0 N Citosina NH, 5' HO Guanina 3' 2 3' 5' N NH OPer studiare la denaturazione del DNA sono stati messi a punto metodi che permettono di seguire la denaturazione del DNA in soluzione misurando l'assorbimento della luce ultravioletta
2,0 spettrofotometro DNA denaturato 1,5 Assorbanza 1,0- 0,5 Thermo DNA nativo 0 200 220 240 260 280 300 Lunghezza d'onda (nm)
DNA e RNA, non assorbono radiazioni elettromagnetiche nell'intervallo di lunghezza d'onda della luce visibile (sono soluzioni trasparenti), la luce ultravioletta (caratteristico picco 260 nm) SÌ OD (Optical Density, assorbanza A) viene comunemente usata in lab per determinare la concentrazione del DNA/RNA in soluzione.
- una lettura di 1 OD 260 = 50µg/mL di dsDNA
- 1 OD 260 = 40ug/ml di RNA
- 1 OD. 260 = 33µg/mL di ssDNA
OD aumenta del 40% se DNA viene denaturato. Dovuto alle basi più mascherate nella struttura a doppia elica che non nei singoli filamenti
Temperatura di Melting (Tm)
temperatura a cui avviene metà denaturazione. dipende da vari parametri legati alla soluzione in cui facciamo avvenire la denaturazione una bassa forza ionica e/o un alto pH della soluzione acquosa destabilizzano la doppia elica; un'alta forza ionica e un pH neutro la stabilizzano, per potere confrontare la Tm di diverse preparazioni di DNA e renderle comparabili i risultati ottenuti in diversi laboratori si esegue la misura della Tm in condizioni standardizzate (NaCl 0,15 M, Na citrato 0,015 M, pH 7,0).
a parità di condizioni differenti, preparazioni di DNA con composizione in basi diversa presentano curve di denaturazione diverse. 100 NaCI 150 mM Na citrato 15 mM Assorbanza a 260 nm aumento relativo (%) Poli (AT) 50 DNA naturale 42% GC Poli (GC) Tn 0 20 40 60 80 100 120 Temperatura (℃)
dipende dal tipo di composizione del DNA (ricco di C-G? ) Tm= 4(G+C)+2(A+T) 4
Riassociazione del DNA
La denaturazione del DNA è un processo reversibile. riassociazione dei filamenti di DNA (annealing). si può formare anche una struttura a doppia elica ibrida tra emielica DNA e di RNA complementari o tra due emieliche di DNA (ibridazione) a temperature relativamente alte e abbastanza vicine alla Tm avremo una riassociazione non molto efficiente ma sicuramente 'stringente"
A B Nucleic Acid Hybridization DNA 5 3' RNA 3 5' DNA-RNA Hybridization GAUC DNA-RNA Hybrid 5' 3 3 5' GAUC AAAAAAAAAAAAAAAA TTTTTTTTTTTTTTTT CTGCTACGACATAGGT GACGATGCTGTATCCA Frammentazione AAAAAAAAAAAAAAAA TTTT TTTT TTTT TTTT CTGC TACG ACAT AGGT G ACG ATGC TGTA TCCA Denaturazione ATGC AGGT ACAT CTGC I Riassociazione AAAA TTTT AAAA TTTT TACG ATGC CTGC GACG AAAA TTTT AAAA TTTT AGGT TCCA ACAT TGTA GACG TTTT AAAA TTTT AAAA TGTA TACG TCCA AAAA AAAA TTTT TTTT 5
EtBr molecules -- - Come produciamo questi frammenti Che si vedono nel gel e in membrane con sonde marcate? DNA Probe DNA hybridization HD Animation https://youtu.be/3LtoKv-XjCo 7
Enzimi di Restrizione (ER)
Biochimicamente ER fanno parte delle endonucleasi provocano delle rotture interne nel dsDNA in corrispondenza di particolari sequenze nucleotidiche, ER entrati nella pratica corrente delle tecnologie del DNA ricombinante. I ricercatori avevano visto che un batteriofago determina la lisi, morte, di cellule batteriche infettate dal virus con percentuali molto diverse da un ceppo all'altro di batteri. restrizione dell'ospite - La capacità di un ceppo batterico di prevenire la replicazione di un virus infettante .
Phage REase MTase Methylated recognition site Recognition site D Ocr Phage T7 MTase MTase 1 EcoRI Phage DNA Restriction enzyme Bacterial chromosome protection by methylation AT TA TA AT A T CRI T restriction enzyme https://youtu.be/lk_Pxht1LM 0?feature=shared® 8
Tipi di Enzimi di Restrizione
ER del I tipo
- riconoscono specifiche sequenze di nucleotidi, ma tagliano il DNA in posizioni non specifiche a una certa distanza dalla sequenza riconosciuta. Poiché il sito di taglio non corrisponde a una specifica coppia di basi, non sono quasi mai utilizzati nelle tecnologie del DNA ricombinante. o NNNN
ER del II tipo
- riconoscono specifiche sequenze di nucleotidi, tagliano il DNA all'interno delle specifiche sequenze, chiamate siti di taglio. La sequenza di riconoscimento per la maggioranza degli ER del II tipo (ne esistono circa 300) è lunga 4-6 nucleotidi, ma esistono ER "rare cutter" che riconoscono sequenze più lunghe.
5'- GAATTC CTTAAG -3' Eco RI 5'- 3'- G AATTC -3' 3'. -5' Digest CTTAA G -5' - 5' sticky-ends la frequenza di taglio di un ER è legata alla lunghezza della sequenza riconosciuta dallo specifico ER 1 5'- CCCGGG -3 Smal 5'- CCC GGG 3'- GGGCCC -5' Digest 3'- GGG -3 CCC -5 blunt ends 10
Nomenclatura e Caratteristiche degli ER
- Gli ER prendono nome dall'organismo dal quale vengono isolati.
- Per convenzione si usa un sistema a 3 lettere in corsivo
- Se un ospite particolare ha diversi sistemi di restrizione-modificazione, questi vengono identificati con i numeri romani (es. HaeI, HaeII, HaeIII).
- Le lettere addizionali indicano un particolare ceppo batterico da cui è stato ottenuto l'enzima (ad es.Bglll -> Bacillus globigii; EcoRI -> Escherichia coli RY13; Sma, Serratia marcescens, HaeIII, Haemophilus aegyptius bacteria ecc).
- la probabilità che sul DNA si trovi una qualsiasi, ma specifica sequenza di 4 paia di basi (pb) è di (1/4)4, cioè 1 volta ogni 256 nucleotidi;
- se la sequenza riconosciuta è lunga 6 pb si troverà statisticamente sul DNA 1 volta ogni 4096 pb (1/4)6.
- enzimi che riconoscano sequenze di 8 pb o più taglieranno il DNA molto raramente (rare cutter).
5'- GAATTC CTTAAG -3' Eco RI 5'- 3' G CTTAA AATTC -3' 3' -5' Digest G -5' 1 5' sticky-ends 5'- CCCGGG -3 Smal 5'- CCC GGG -3 3'- GGGCCC -5' 3'- GGG CCC -5 Digest blunt ends Serratia marcescens 12
Digestione del DNA e Elettroforesi
DIGESTION OF DNA NONSPECIFIC BY RESTRICTION ENDONUCLEASE DNA FRAGMENTS dreamstime 10-100 ul dreamsti dreams dreamstime Seam/time reamstime 80 44067505 imgflip.com Download from Dreamstime.com This watermarked comp image is for previewing purposes only. Luchschen | Dreamstime.com 13
https://youtu.be/kjJ56z1HeAc Agarose Gel Electrophoresis of DNA fragments amplified using PCR - - alamy TH TTL Western blot acrilammine Roti8-Nylon plus Southern blot - DNA Northern blot - RNA Example 1: Using DNA probes to search a match on an organism's DNA Filter paper Gel 1 2 3 4 5 6 7 8 9 -- (a) kb 23-13 9.42 6.56 4.36 2.32 2.03 1.35 1.08 0-87 0.60 14
Western blot - proteine - gel acrilammide invece che agarosio Sealable plastic bag Labeled probe =FLUORESCENZA
Bromuro di Etidio (EtBr)
"Il bromuro di etidio (EtBr) è un colorante fluorescente intercalante (non radioattivo, un composto eterociclico aromatico. Il nome chimico dell'EtBr è 3, 8-diammino-5-etil-6- fenilfenantridinio) del DNA usato per visualizzare il DNA durante l'elettroforesi su gel. - EtBr molecules Viene aggiunto al tampone di corsa o nel gel e si lega intercalando tra le coppie di basi del DNA. Quando il gel di agarosio viene illuminato con luce UV 15
Il bromuro di etidio molecola intercalante di acidi nucleici (DNA e RNA) comunemente utilizzata nella elettroforesi su gel di agarosio. Quando DNA esposto ai raggi ultravioletti, emette fluorescenza che si intensifica se intercalato nel DNA. con bromuro di etidio, è possibile evidenziare lo stato della migrazione del DNA all'interno del gel. bromuro di etidio migra dal polo positivo al polo negativo del campo elettrico, in direzione opposta a quella del DNA. ethidium bromide (marked red) 1 1 H2N -NH2 1 - ) N Br -CH3 intercalates between base pairs 16
Sonde di DNA per Identificazione e Analisi di Sequenze
sonde di DNA per identificare e analizzare sequenze uso della reazione di riassociazione di filamenti complementari viene fatto in tecniche in cui viene utilizzata una breve sequenza sonda (probe) per evidenziare e studiare sequenza complementare in una complessa preparazione di DNA (o RNA). must first be isolated from bodily samples through chemical extraction methods before a DNA probe can be used to identify pathogens. Denatured SSDNA DNA probes kb gene of interest 23.13 9.42 6.56 + 4.36 I I 2.32 I 2.03 1 I - 1 Isolate DNA from body fluid sample. 2 Denature DNA sample and combine with DNA probes. Probes are complementary to the gene of interest and labeled with a molecular beacon. 3 DNA probes will bind to the gene of interest if it is present in the DNA sample. 1.08 --- 0.87 -- ad to naufica T am illiintentan han. ha non ha · Atomo radioattivo · Una "etichetta" fluorescente (a) 1 2 3 4 5 6 7 8 9 DNA sample Marcatura * fluorescente/radioattiva 1.35 I 0.60 17
Microarray
A B Purificazione dell'mRNA mRNA Preparazione del microarray 1 Trascrittasi inversa marcatura cDNA fluorescente Į Ibridazione su microarray Analisi dei segnali mediante laser C I microarray (A) un robot automaticamente prepara microarray: un supporto su cui sono allineati in maniera ordinata migliaia di sonde (DNA) rappresentativi dei diversi geni. (B) RNA estratto da cellule usato come stampo e marcato a fluorescenza (eventualmente PCR) ma prima cDNA 2 mRNA Reverse transcriptase Single Stranded DNA DNA polymerase 88 Complementary DNA (double stranded) (C) microarray incubato con i cDNA, lavato e analizzato per i segnali di fluorescenza. profilo d'espressione genica. 18
Confronto tra Preparazioni di RNA con Microarray
A B https://youtu.be/6ZzFihESjp0 DNA microarray Confronto tra due preparazioni di RNA mediante microarray. RNA estratti dai campioni cellulari A e B usati come stampo per la sintesi di cDNA fluorescente mediante RT-PCR utilizzando due fluorofori diversi, verde A e rosso B. I due cDNA mescolati in un'unica soluzione nella quale viene poi immerso e incubato il microarray. L'analisi dei segnali di fluorescenza mostra segnali verdi e alcuni rossi, che corrispondono a geni espressi di più nelle cellule A e nelle cellule B, rispettivamente. I più numerosi segnali gialli corrispondono ai geni espressi più o meno allo stesso livello nei due campioni cellulari. trattate Non trattate 1 Purificazione dell'mRNA mRNA mRNA 1 Trascrittasi inversa e marcatura cDNA fluorescente cDNA fluorescente Mescolare i due cDNA Ibridazione su microarray Analisi dei segnali La C 19