Lezione 1: Concetti fondamentali di biologia molecolare per universitari
Documento di Lezione 1 sulla biologia molecolare. Il Pdf esplora i concetti fondamentali della biologia molecolare, inclusa la distinzione tra cellule procariotiche ed eucariotiche, la struttura del DNA e RNA, e il processo di trascrizione, utile per studenti universitari di Biologia.
Mostra di più51 pagine
Visualizza gratis il Pdf completo
Registrati per accedere all’intero documento e trasformarlo con l’AI.
Anteprima
Procarioti vs Eucarioti
PROCARIOTICA vs EUCARIOTICA: hanno compartimentazione delimitate da membrane (che avvolgono il materiale genetico, solo negli eucarioti).
Citoplasma Nucleoide Ribosomi 0,5 um Parete cellulare Membrana citoplasmatica Ribosomi Membrana nucleare Reticolo endoplasmatico Citoplasma Membrana citoplasmatica Mitocondrio Apparato del Golgi Nucleo Nucleolo 10 um
La compartimentazione eucariotica è la causa della specializzazione dei metabolismi, all'interno di organelli dedicati. Dall'interno: una membrana citoplasmatica, una parete cellulare, um nucleoide (materiale genetico), il citoplasma (in ambiente acquoso) e i ribosomi (numerosi, producono proteine).
Trasmissione dell'informazione genetica
Gli acidi nucleici trasmettono l'informazione genetica, si è scoperto in seguito a degli esperimenti.
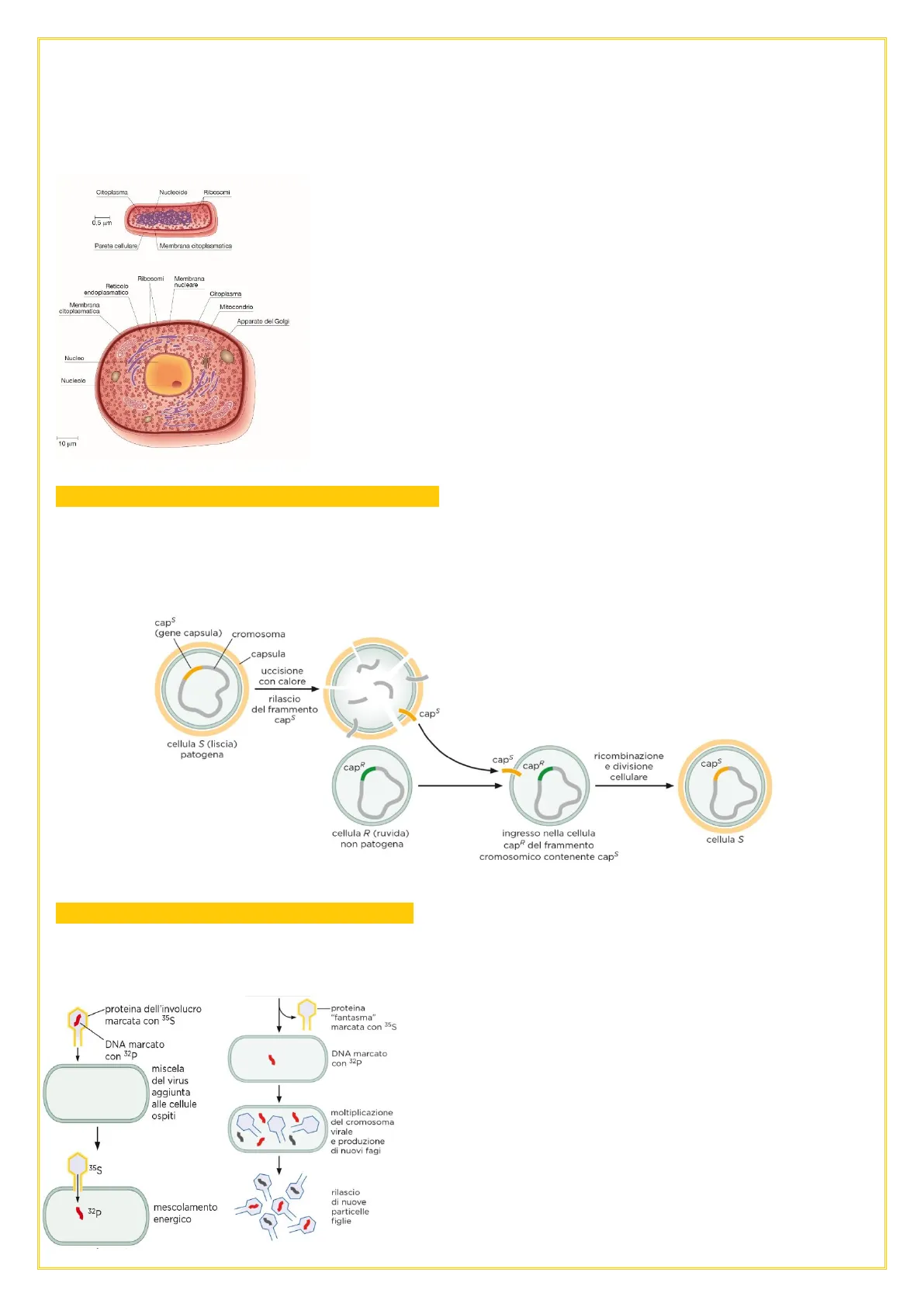
Esperimento di Griffith
Nei primi anni 30, Girflield (?), prese batteri incapsulati, virulenti, vivi ed ha iniettato questi batteri all'intenro di un topo, causandone la morte. Prese allora batteri non incapsulati, non virulenti ma vivi, iniettandolo in un altro topo, e continua a vivere. Prese i batteri incapsulati virulenti vivi sottoponendo al calore, uccidendoli, e li iniettò nel topo che continuò a vivere. I batteri non incapsulati, virulenti e vivi uccisi al calore uniti a batteri non virulenti vivi: il topo muore (i batteri morti da virulenti contenevo l'informazione virulente). Mescolando il DNA con batteri non virulenti non incapsulati e vivi, escono batteri incapsulati virulenti portando alla morte del topo.
Esperimento di Avery, MacLeod e McCarty
ESPERIMENTO DI AVERY, MACLEOD E MCCARTY: cellule con all'interno informazione per la capsula, il rivestimento; questi batteri venivano definiti S (da smooth, liscio) perchp l'incapsulamento gli conferiva l'aspetto di essere lisce e la patogenicità. Le altre, con informazione R (ruvida, rugose) non incapsulate e non patogene. Mescolando cellule S uccise al calore e cellule R si verificava che le cellule R acquisivano l'informazione contenute nel gene capsula (portava informazione per la codifica della capsula), del rivestimento: la cellula da ruvida diventava liscia. Il gene capsula, capS, con l'isolamento del DNA e l'aggiunta delle cellule non patogene le trasformava (trasformazione: inserire dna all'interno) in patogene lisce.
caps (gene capsula) cromosoma capsula uccisione con calore rilascio del frammento caps caps cellula S (liscia) patogena caps capR ricombinazione e divisione cellulare caps cap® cellula R (ruvida) non patogena ingresso nella cellula cap® del frammento cromosomico contenente caps cellula S
Esperimento di Hershey e Chase
ESPERIMENTO DI HERSHEY E CHASE TAGO T2: aiuta a capire la molecola depositante dell'informazione. Utilizzarono dei batteriofagi marcati (modificati, rendendoli "visibili" mediante molecole radioattive con isotopi radioattivi dello zolfo e del fosforo perchè lo zolfo 35 può essere incamerato all'interno della macromolecola della meteorina e cisteina, così che produca proteine incamerando lo zolfo 35 nelle proteine stesse; il fosforo 32 è radioattivo e contenuto nel DNA). Si potevano creare batteriofagi con proteine e DNA differenti: con fosforo 32 e zolfo 35.
proteina dell'involucro marcata con 35S proteina "fantasma" marcata con 35s DNA marcato con 32p 5 DNA marcato con 32p miscela del virus aggiunta alle cellule ospiti moltiplicazione del cromosoma virale e produzione di nuovi fagi 35S 5 32p mescolamento energico rilascio di nuove particelle figlie
Il batteriofago si avvicina al batterio e trasferisce il materiale genetico all'interno; la parte esterna del batterio in seguito a un mescolamento energico si stacca: il DNA del batteriofago entra nel batterio, le proteine (gialle nell'immagine perché è marcato) e la parte radioattiva rimane fuori. Quindi la parte proteica del batterio rimane esclusa, dentro c'è il DNA rosso (marcato con fosforo 32); il DNA che si inserisce e riproduce nell'ospite contiene informazione anche per le proteine e verranno prodotte particelle virali figlie con materiale genetico nuovo non radioattivo e quelle con incorporato il materiale genetico. Si formerà DNA radioattivo con proteine radioattive con 32p mentre con 35S si formeranno cellule di virus con DNA non radioattivo.vecchio vecchio A C T vecchio nuovo nuovo vecchio
Struttura del DNA
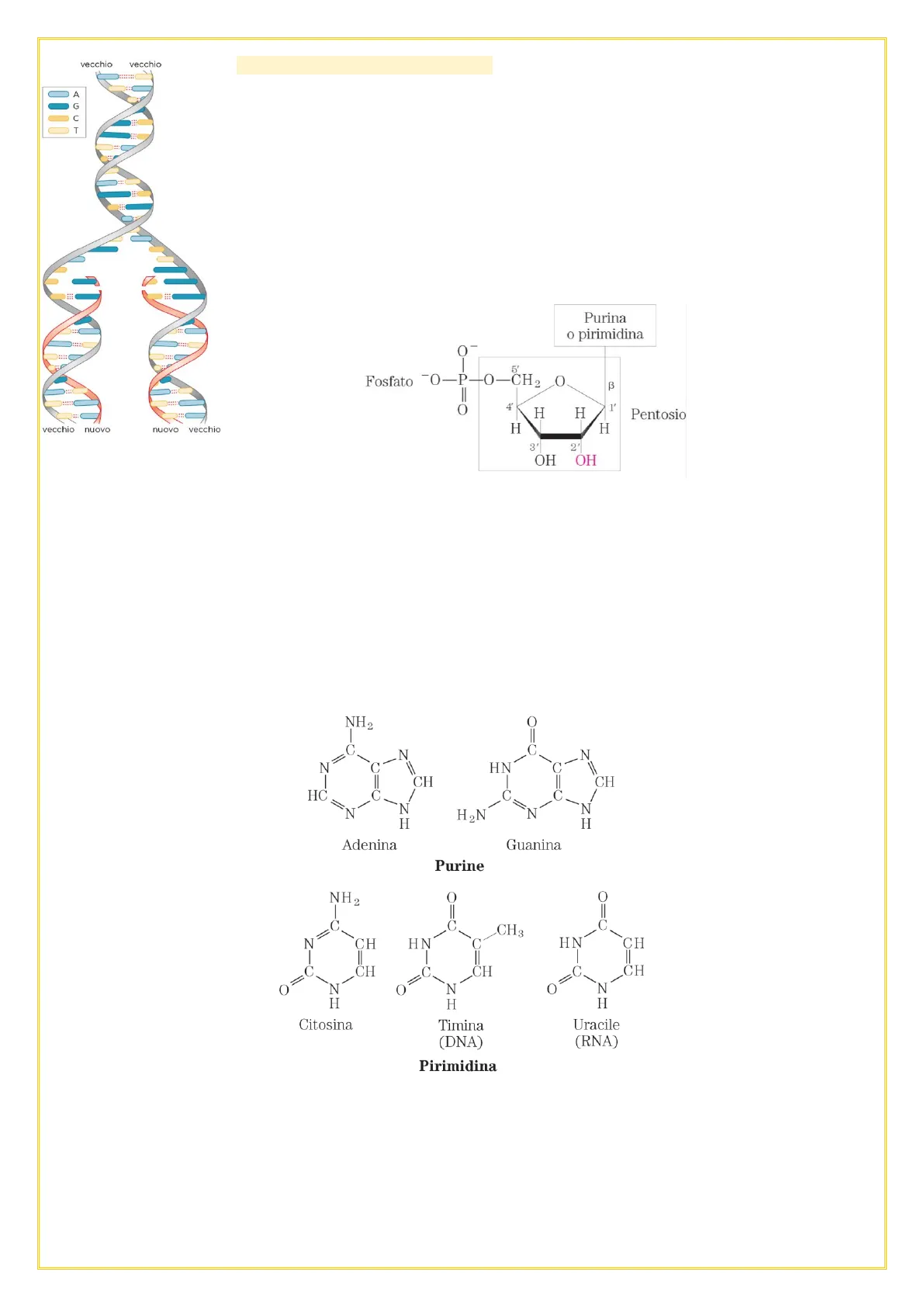
Purina o pirimidina O 5' Fosfato 0-P-O-CH2_O II O 4 1' H H Pentosio H H 3' OH OH
Lo zucchero è un furanosio, cioè un ciclo a cinque. Le purine (adenina o 6-amino-purine e guanina o 2-amino-6-oxy purine), le piramidine (citosina o 2-oxy-4-amino pyramidine, timina nel dna o 2-oxy-4-oxy 5-methyl pyramidine e uracile nell'RNA o 2-oxy-4- oxy pyramidine).
Nel tRNA si trovano: Inosine, Pseudoridina (pseudo in quanto è diversa, perché lo zucchero non ha legame n-beta ma il ribosio è legato al carbonio 5), 7-Methylguanosine e 4-Thiouridine (perché in posizione 4 c'è un doppio legame con lo zolfo).
Chimicamente parlando, le basi in funzione di ph possono esistere in più tautomeri: il tautoremismo chetoetomico. Nel lattame (gli elettroni di legame vanno all'ossigeno e da chetone), nel lattime (diventa enolo, tautomerismo chetoenolico) e nel doppio lattime.
NH 2 O II C N HN CH C C N HON N H Guanina Purine NH2 0 O II C CH3 N CH HN C HN CH C CH C CH 0 = N O N O N H H H Citosina Timina (DNA) Uracile (RNA) Pirimidina N Il CH HC C N N H Adenina
NEL 53 PRIMA IMMAGINE DEL DNA ai raggi X grazie a Rosalind Franklin confermando l'ipotesi che il DNA fosse elicoidale. La doppia elica quando c'è sintesi di DNA nuovo (immagine con dna vecchio e nuovo): * struttura nucleotide importante all'esame* il DNA è un acido nucleoico fatto da nucleotidi A, G, C, T rappresentano le basi che contribuiscono a costittuire i nucleotidi (struttura chimica: una base o purina o pirimidina, legata tramite legame N-beta-glicosidico ad uno zucchero pentosio con 5 atomi di C *immagine struttura*, è un furano. Nucleoside: base più zucchero). La posizione 2' è contenuto nello zucchero RIBOSIO, che si ritrova nei ribonucleotidi (che costituiscono l'RNA); DNA riossidublenocleico: il deossi vuol dire che manca l'ossidrile.
LE BASI: purine o pirimidine. Sono eterociclici, quindi cicliche con atomi diversi dal CARBONIO, infatti hanno atomi di azoto. La differenza sta el fatto che le seconde sono fatte di un solo anello, mentre le prime hanno due anelli condensati, un ciclo a 6 e uno a 5. Sono molecole aromatiche (hanno elettroni che risuonano in tutta la molecola).
C C
Stabilità dell'informazione genetica
INFORMAZIONE DEVE ESSERE STABILE: nel tempo deve rimanere costante. Nel corso dell'evoluzione cambia, spontaneamente perché possono esserci modificazioni delle basi: questi processi coinvolgono il DNA. Se la variazione di informazione porta ad un miglioramento, del fenotipo, delle caratteristiche acquisite; se questa variazione è tossica, succederà che la cellula che ha subito la trasformazione morirà.
Le reazioni serviranno a contrabbilanciare la risposta ad eventi spontanei. Una delle variazioni spontanee a cui vanno incontro le basi è la DEAMMINAZIONE (perdita di un gruppo amminico) ad ex quello della citosina porta ad un uracile. Questa trasformazione che si verifica in 1 su 10^7 in 24 ore, cioè 100 volte al giorno una citosina viene convertita in uracile.
Le regole dell'appaiamento tra basi: avvale dell'esperimento di Chargaff. L'uracile, contenuto nell'RNA, quando una citosina diventa tale, i sistemi di riparazione devono individuare quale appaiamento è scorretto.
La depurinazione (con purine come basi) significa perdita di una base. Una guanosina (glucoside) ad esempio, che ogni 24h, 1 su 10^5, in cui si rompe il legame n-beta e si rilascia la guanina e si ha un residuo apurinico.
Nucleoside (base + zucchero), nucleotide (+ fosfato).
Regola di Chargaff
DATI DELLA FORMULAZIONE DELLA REGOLA DI CHARGAFF: nel 1949, prima della struttura del DNA, analizzò il contenuto in vari genomi di specie diverse, sul rapporto tra le varie basi dei genomi delle varie specie.
Legame fosfodiestereo
IL LEGAME FOSFODIESTEREO: coinvolge l'ossidrile in 3' di uno zucchero del primo nucleotide, a cui seguono base zucchero e fosfato fino al prossimo 5' del nucleotide che sta entrando. La catena ha un verso, e il legame che tiene uniti i due rubonucleotidi è sempre un legame fosfodiestereo.
K-2,8Å-> CH 3 5' 3 H H -0 Timina C G N IHIH-N C-N H-C C-1' + Adenina T ~C. C. N H C-1' -11,1 Å -2,9 Å-> H H C Citosina N -N H-C C-1' 1 2 N-HILIO C-1' H K-2,9Â-> -10.8 Å G 3' 5'
Il DNA è fatto di due filamenti ANTIPARALLEILI, uno orientato a 5' 3' e l'altro a 3' 5'. C e G sono legate da 3 legami idrogeno mentre A e T da 2. L'appaiamento dei rispettivi, per allontanare due filamenti complementari di DNA, non si può non tenere in considerazione che ci vuole più energia per rompere il terzo legame, tramite contributo termico. La distanza tra H-O o H-N: intorno ad 3 Armstrong.
L'informazione è contenuta nel DNA in quanto molecola molto più stabile del RNA; anche se alcune specie possono avere informazioni nel RNA, come nei batteri.
In soluzione acquosa, in ioni H-, si può strappare il protone: gli elettroni si possono attaccare intramolecolarmente al fosfato del ponte fosfodiestereo portando all'accorciamento del RNA, in conseguenza della formazione del composto 2',3'- monofosfato ciclico.
Forme del DNA
Il DNA può esistere in diverse forme, non tutte fisiologiche: forma A, B (quella più vicina alle forme fisiologiche) e Z, le prime due destrorse, l'ultima sinistrorsa e sono in ordine di larghezza. I giri dell'elica costituiscono la variabilità delle coppie di basi per ogni giro. Le basi si trovano impilate una sull'altra e la distanza di queste la si misura con Armstrong: 3,4 ad esempio per la forma B.
Il DNA a tripla elica è un momento della vita del DNA in cui si afferma questo fenomeno.
(b) A DNA (c) Z DNA (d) Triple-Helical DNA (a) BDNA wÚ 3.4 nm H k-3,0 Å-> N-HIIIN GIII C Guanina A 10 -3,0 Å-> C 0
AProtein Data Bank: PDB è una banca dati di strutture tridimensionali di proteine ma anche di acidi nucleici. Il codice identificativo: 1BWG, in cui sono depositate le coordinate atomiche. L'avvolgimento è dato da due scanalature: una minore e una maggiore: sono la conseguenza di ciò che si instaura.
I 3,4 ÅR Scanalatura minore 36 Å Scanalatura maggiore 20 Å (a) (b) (c)
Denaturazione e riassociazione del DNA
Il DNA: la separazione dei due filamenti è un evento non immediato, in vitro sottoposto al calore. La denaturazione e la riassociazione: dato dal riscaldamento, dalla sua presenza e della sua assenza. Rapporto di percentuale di denaturazione e temperatura: temperatura di Melting, esprime la denaturazione avvenuta di almeno il 50%. Più è elevato il livello di guanina e citosina nel DNA, più elevata è la percentuale di denaturazione.
TN= 4℃ * (G+C) + 2℃ * (A+T)
La bolla di duplicazione: si forma in punti specifici e dipende dall'ORIGINE DI REPLICAZIONE: ori-ci
100 DNA a doppia elica Denaturazione Riassociazione (annealing) Denaturazione (%) 50 tm tm 0 75 80 85 (a) Temperatura (℃) Separazione delle catene Associazione delle catene mediante appaiamento delle basi & Catene separate di DNA con una struttura ad avvolgimento casuale G + C (% dei nucleotidi totali) 80 60 40 20 0 60 70 80 90 100 110 (b) tm (C) DNA parzialmente denaturato 100