Nucleotidi: funzioni, GTP, proteine G e sintesi proteica
Slide dall'Università sui Nucleotidi. La Pdf, utile per studenti universitari di Biologia, approfondisce i nucleotidi, il GTP, le proteine G, la trascrizione, la maturazione e il tRNA, con diagrammi esplicativi.
Mostra di più59 pagine

Visualizza gratis il Pdf completo
Registrati per accedere all’intero documento e trasformarlo con l’AI.
Anteprima
Nucleotidi e Funzioni
Nucleotidi
Oltre ad essere componenti degli acidi nucleici (DNA e RNA) i nucleotidi possono svolgere altre
importanti funzioni. Es.
- La maggior parte dell'energia utilizzata dagli esseri viventi deriva dall'adenosina
trifosfato (ATP) - La guanosina trifosfato (GTP) è un nucleotide che, oltre a fornire energia per la sintesi
proteica, si lega d un gran numero di proteine ("proteine G") e agisce da interrutore
per attivarle. - Proteine G monomeriche
- Proteine G trimeriche.
- L'AMP ciclico (cAMP) è un ribonucleotide speciale che è essenziale per l'azione
ormonale e per il trasferimento di informazione nel sistema nervoso.
GTP e Proteine G
GTP e "Proteine G"
- Funzionano da interrutori molecolari. Quando legano
la guanosina trifosfato (GTP) si attivano ("on") e quando
legano la guanosina difosfato si disattivano ("off"). - Le proteine G regolano enzimi metabolici, canali ionici,
trasportatori di membrana, e altre componenti delle
attività cellulari, controllando la trascrizione, motilità,
contrattilità e secrezione che, a loro volta, regolano
funzioni sistemiche quali lo sviluppo embrionale,
l'apprendimento, la memoria e l'omeostasi.
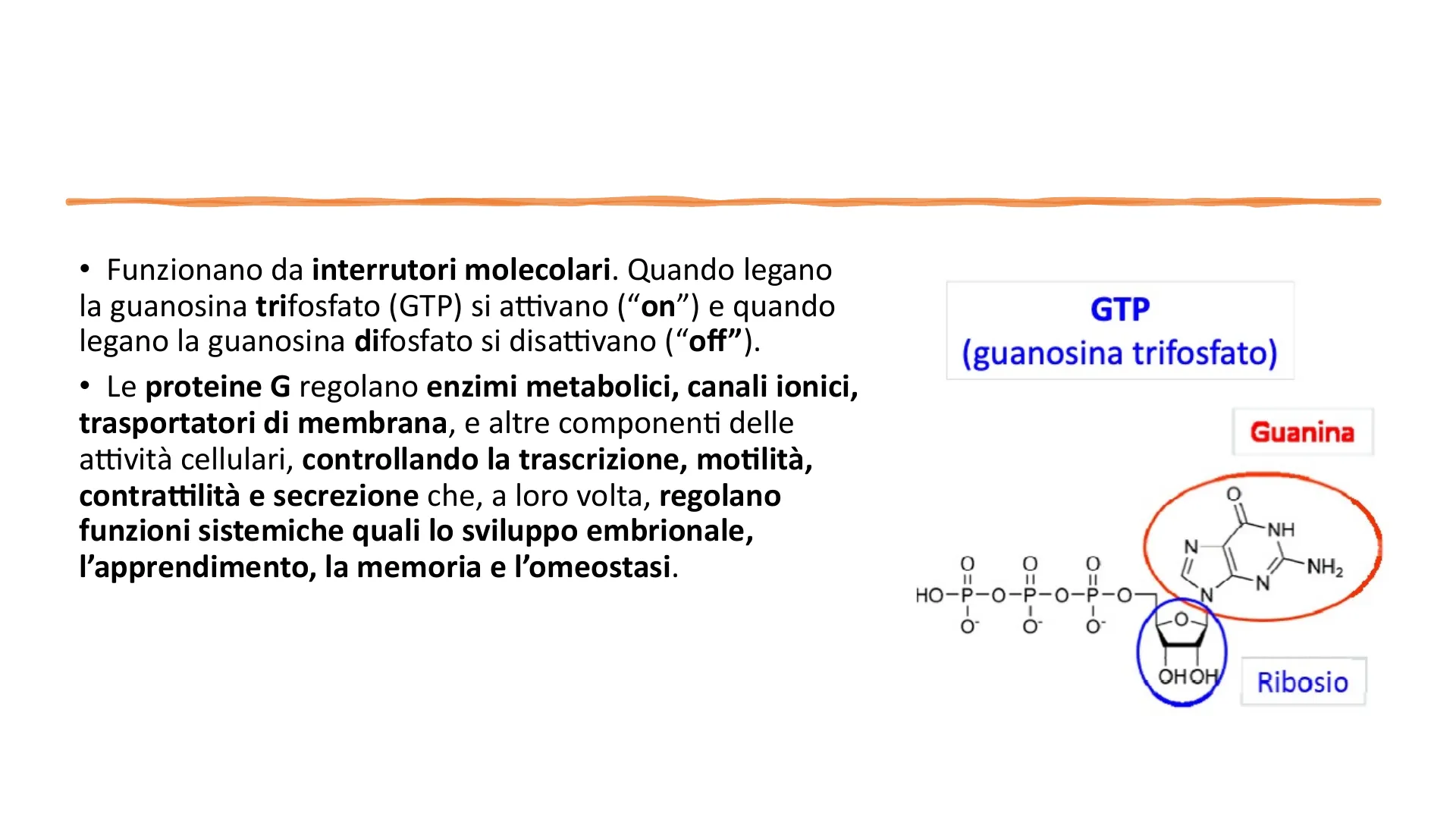
Struttura della Guanosina Trifosfato (GTP)
GTP
(guanosina trifosfato)
Guanina
O
-NH
N
0
C
0
NH2
N
HO-P-O-P-O-P-O-
N
1
O=0-0
-O
OHOH
Ribosio
AMP Ciclico (cAMP)
AMP ciclico (cAMP)
- L'adenosina monofosfato ciclico (AMP ciclico o cAMP)
è un metabolita delle cellule prodotto dall'enzima
adenilato ciclasi a partire dall'ATP. È un importante
"secondo messaggero" coinvolto nei meccanismi di
trasduzione del segnale all'interno delle cellule viventi
in risposta a vari stimoli, come quelli indotti dagli
ormoni glucagone o adrenalina, che non sono in grado
di attraversare la membrana cellulare. La sua principale
funzione consiste nell'attivazione di protein chinasi A
(PKA) per regolare il passaggio transmembrana di calcio
attraverso i canali ionici oppure tramite cascata porta
all'aumento del glucosio disponibile come conseguenza
di una degradazione del glicogeno.
Struttura dell'AMP Ciclico
NH2
N
N
N
N
O
O
HO
P
O
O
OH
Accettori Intermedi di Elettroni: NAD+, NADP+
Accettori intermedi di elettroni: NAD+, NADP+
NAD
NH2
N.
EN
H2N
N
N
O
C-O-P-O-P-O-C
H2
Q=0-0
O=0-0
H2
HỒ
OH
HO
OH
NAD+
+
2[H]
H
O
C-NH2
N
Nicotinamide
(oxidized form)
O
CH2
o
O=P-O"
N
N
O
H
H
HO
OH
O=P-O"
NH_
O
CH2
N
N
H
N
N
O
NAD+ in mitochondria
& citosol
HO
OH
HO
O-P-OH
HO
2 e + 2 H+ -
2 e" + H+
Dehydrogenase
oxidoreductase
NADH
O
H
H
C-NH2
+
Oxidation of NADH
N
Nicotinamide
(reduced form)
O
NADP
NH,
N
N
HÂN
0=0-0
O=0-0
0
C-O-P-O-P-O-C-
H2
H2
O=0
H
& citosol
エ ––▶ 主
Reduction of NAD+
N
O
niacin
vitamin - B3
O
O
Entrambi sono accettori di ioni H+ e si trasfomrano
nella loro forma ridotta NADH e NADPH quando
un substrato va incontro a ossidazione
*released into surrounding solution
H+MM
HHO
:H-
NH2
o-
0
o=P-O
O=P-O-
N
O
OH OH
NH2
->
NH2
+ H+
N
N
SN
N
0=P-O-
o=P-O.
N
N
-0
0
-0
OH OH
OH OH
NAD+
NADH
+ H+
NH2
N
OH OH
N
N
Composizione dei Nucleotidi
NUCLEOTIDI
Basi
Zuccheri
Gruppi fosfato
Altri componenti
| ATP | = | Adenina | + | Ribosio | + | 3 | |
| ADP | = | Adenina | + | Ribosio | + | 2 | |
| CAMP | = | Adenina | + | Ribosio | + | 1 | |
| NAD | = | Adenina | + | 2 Ribosio | + | 2 | + Nicotinamide |
Polimeri Nucleotidici
POLIMERI
NUCLEOTIDICI:
| DNA | = | A,G,C,T | + | + | Deossiribosio | + | + | 1 per nucleotide |
| RNA | = | A,G,C,U | Ribosio | 1 per nucleotide |
Adenosina Trifosfato (ATP)
Adenosina trifosfato (ATP)
L' ATP è il più abbondante
trasportatore energetico
nelle reazioni cellulari.
La rottura del legame
fosfoanidrinico libera
grandi quantità
energetiche e il gruppo
fosfato può rimanere
libero o essere trasferito
ad altre molecole.
ATP
legame fosfoanidridico
O
O-
O
ADENINA
0-P-O-P-O-P-O-CH2
II
RIBOSIO
Degradazione
nutrienti
+ H20
H20 -
Biosintesi,
Movimenti,
ecc
O
-
ADENINA
H++ +0-P-OH
+
-O-P-O-P-O-CH2
Ö
Ö
O
fosfato
inorganico (Pi)
RIBOSIO
ADP
Macromolecole Biologiche e Funzioni
Le macromolecole biologiche e
le loro funzioni
DAL DNA ALLE PROTEINE
-trascrizione-
(parte Iº)
Espressione Genica
Espressione genica
Gene
Chemical nature
DNA
Sequence of nucleotides
1
Transcription
RNA
Sequence of nucleotides
1
Translation
Polypeptide
Sequence of amino acids
Metabolites
Sintesi dell'RNA e Proteica
gene
DNA
5'
3'
3'
5'
SINTESI DELL'RNA
TRASCRIZIONE
nucleotidi
RNA
5'
3ʹ
SINTESI PROTEICA
TRADUZIONE
PROTEINA
H2N.
COOH
amminoacidi
Espressione
genica
Processo di Splicing
EXON 1
EXON 2
DNA
INTRON
Transcription
NUCLEUS
5'
INTRON
3'
Pre-mRNA
INTRON
Splicing
5'
3'
mRNA
Transport
tRNA
5'
3'
Ribosome
Translation
CYTOPLASM
Protein
Trascrizione del DNA in RNA
TRASCRIZIONE
La trascrizione è la sintesi di una molecola di
RNA la cui sequenza di basi è complementare a
quella del filamento stampo.
filamento codificante
DNA
5
3
3'
5'
filamento stampo
TRASCRIZIONE
5'
'3'
RNA
RNA polimerasi
5'
3'
5'
filamento stampo
di DNA
trifosfati
del ribonucleoside
in entrata
5'
sito attivo
canale di trifosfati
della polimerasi del ribonucleside
trascritto di RNA
appena sintetizzato
L'enzima RNA polimerasi
trascrive il DNA in RNA.
Nelle cellule eucariotiche
esistono 3 diverse RNA
polimerasi (I-II-II) attive
nella trascrizione di tipi
differenti di geni.
Filamento di DNA Trascritto
Un filamento di DNA è trascritto in RNA
DNA
Filamento codificante
Filamento stampo
5' TACGCGGTACGGTCAATGCATCTACCT
3' ATGCGCCATGCCAGTTACGTACATGGA
TRASCRIZIONE
La sequenza dell'RNA è
complementare al filamento stampo
equivalente al filamento codificante
5'UACGCGGUACGGUCAAUGCAUCUACCU
RNA
Trascritto di RNA
Filamento CODIFICANTE:
-orientamento 5'-3'
-stessa sequenza nel TRASCRITTO di RNA (U al posto di T)
filamento STAMPO o TEMPLATE
-costituisce lo STAMPO (viene usato per appaiare le basi)
-è complementare al trascritto
Classi di RNA
Classi di RNA
UNIVERSALI
mRNA: codifica le proteine
tRNA: trasporto di aminoacidi per traduzione
rRNA: costituenti del Ribosoma
RNA REGOLATORI
snRNA: (piccoli RNA nucleari):
assistono durante lo splicing (spliceosoma)
snoRNA modificano gli rRNA
small RNA: microRNA, piwiRNA, siRNA ...
RNA non codificante a funzione regolatoria
Long non coding RNA (IncRNA) ...
Xist (X inactive specific transcripts)
Crispr RNA: tracrRNA, crRNA
Tipi di RNA nella Sintesi Proteica
La sintesi proteica usa tre tipi di RNA
L'mRNA ha una sequenza di basi che
rappresentano la proteina
Dimensioni variabili
da 500 a 10 000 basi
Il tRNA è un piccolo RNA
con un'estesa struttura
secondaria e dimensioni
da 74 a 95 basi
Gli RNA ribosomali principali
hanno un'estesa struttura
secondaria e si associano a
proteine per formare il ribosoma
Dimensioni da 1500 a 1900
(rRNA più piccolo) e da 2900
a 4700 (rRNA più grande)
Struttura del tRNA
3'
A
Sito di legame
con l'amminoacido
C
C
5'
...
tRNA
...
...
...
Ansa T
(ansa 3)
...
....
...
...
Ansa D
(ansa 1)
-
C-
....
Ansa
variabile
Ansa
dell'anticodone
(ansa 2)
C
Anticodone
GUCCAG
GAG
GUADAG
mRNA
Codone
Codice Genetico e Codoni
SECONDA LETTERA
U
C
A
G
UUU
UAU
Tirosina
UGU
UGC
Cisteina
UUC
C
U
UUA
UCA
UAA
Codoni
di stop
UGA
Codone
di stop
A
UUG
UCG
UAG
UGG
Triptofano
G
CUU
CCU
CAU
CGU
U
CUC
CCC
CAC
CGC
C
C
Leucina
Prolina
CAA
CGA
A
CUG
CCG
CAG
CGG
G
AUU
ACU
AAU
AGU
U
AUC
Isoleucina
ACC
AAC
AGC
C
A
AUA
ACA
AAA
AGA
A
AUG
Metionina
Codone
di inizio
ACG
AAG
AGG
G
GUU
GCU
GAU
GGU
U
GUC
GCC
GAC
aspartico
GGC
C
G
Valina
GCA
GAA
Acido
GGA
A
GUG
GCG
GAG
glutammico
GGG
G
TERZA LETTERA-ESTREMITÀ 3'
La relazione tra
sequenza nucleotidica e
sequenza
amminoacidica è basata
su una serie di regole
che vengono definite
codice genetico.
Le parole del codice
sono i codoni, cioè
triplette specifiche di
basi che corrispondono
a specifici amminoacidi.
PRIMA LETTERA-ESTREMITÀ 5'
Leucina
UCU
UCC
U
Fenilalanina
UAC
Serina
Istidina
Arginina
CUA
CCA
Glutammina
Asparagina
Serina
Treonina
Lisina
Arginina
Glicina
GUA
Alanina
Acido
Tipi e Funzioni di RNA
Nella cellula vengono prodotti diversi tipi di RNA.
| Tipo di RNA | Funzione | |
| RNA messaggeri (mRNA) | Codificano le proteine | RNA pol II |
| RNA ribosomali (rRNA) | Formano il nucleo centrale del ribosoma e catalizzano la sintesi proteica | RNA pol I e III |
| RNA transfer (+RNA) | Fungono da adattatori tra I'mRNA e gli amminoacidi nella sintesi delle proteine | RNA pol III |
| microRNA (miRNA) | Regolano l'espressione genica | RNA pol II |
| Altri RNA non codificanti | Attivi nello splicing, nella regolazione dei geni, nel mantenimento dei telomeri e in molti altri processi | RNA pol II |
Struttura di un Gene
La struttura di un gene
I promotori e i terminatori definiscono l'unità
C
Punto di inizio
Promotore
Terminatore
-35 -10 -1+1 +10
Prossimale
Distale
A monte
A valle
nucleotide +1 del DNA (filamento
codificante):
corrisponde al primo ribonucleotide
presente nel trascritto (start point)
porzione a monte (upstream):
contiene, quasi sempre, il promotore
(numerazione negativa)
porzione a valle (downstream): si trova
la regione codificante (in eucarioti
introni ed esoni)
ed il terminatore (numerazione positiva)
unità trascrizionale: tratto di DNA compreso tra punto di inizio e terminatore
per la RNA polimerasi (può comprendere più geni)
Passaggi della Trascrizione
Trascrizione
https://app.jove.com/it/science-education/v/11581/transcription?playlist=6957188
Passaggi principali della trascrizione
Unità di trascrizione
DNA
5'
in in
3
5'
Promotore
Terminatore
DNA srotolato
(bolla di
trascrizione)
RNA
polimerasi
1 Legame dell'RNA polimerasi
e srotolamento locale del DNA
5'
3'
3'
5'
NTP
2 Inizio della sintesi dell'RNA
PP
1
3'
3'
5
NTP
RNA
3 Allungamento dell'RNA
5'
3
3'
5'
5
RNA
NT
Ulteriore allungamento
5
3
5'
5'
RNA polimerasi
4 Terminazione della sintesi
dell'RNA
5'
3
3
5'
5'
3'
Trascritto di RNA
Appositi segnali sul DNA indicano
all'RNA polimerasi dove cominciare
la trascrizione e dove deve
completarla.
Segnali di Inizio e Terminazione
TATA box
(A)
PROMOTORE
-35
-10
+1
5'
-TAGTGTATTGACATGATAGAAGCACTCTACTATATTCTCAATAGGTCCACG-3'
7 DNA
3'
-
- ATCACATAACTGTACTATCTTCGTGAGATGATATAAGAGTTATCCAGGTGC-5'
sito di
inizio
filamento stampo
TRASCRIZIONE
5'
3'
RNA
AGGUCCACG
(B)
SEGNALE DI TERMINAZIONE
5'
-CCCACAGCCGCCAGTTCCGCTGGCGGCATTTTAACTTTCTTTAATGA-3'
3'
-
GGGTGTCGGCGGTCAAGGCGACCGCCGTAAAATTGAAAGAAATTACT-5'
filamento stampo
TRASCRIZIONE
sito di
terminazione
5'
3'
RNA
CCCACAGCCGCCAGUUCCGCUGGCGGCAUUUU
7 DNA
3'
3'
PP
3
Filmento
stampo
3
5'
Filamento
codificante
1
+
Fattori di Trascrizione e RNA Polimerasi
Nelle cellule eucariotiche le RNA
polimerasi richiedono il concorso di
numerose proteine per iniziare la
trascrizione.
I fattori di trascrizione sono proteine
che si aggregano al promotore
posizionando I'RNA polimerasi e aprono
la doppia elica per esporre il filamento
stampo.
N
A
C
A
A
G
T
A
T
5'
5'
3'
Proteina TBP che si lega al
TATA box distorce la doppia
elica del DNA (in rosso).
Complesso di Pre-inizio
Promotore core
TATA
Start
DNA
TBP
TFIID si lega
alla TATA box
sul DNA.
TFHID
TFILA
TEUB
2 TFIIA e TFIIB
formano il complesso
con TFIID.
TFIIF
RNA polimerasi Il
Dominio
C-terminale
(CTD], "coda"
Il complesso risultante
è legato alla RNA polimerasi
attaccata a TFIIF.
TFIE
TFIIH
4 Il complesso di pre-inizio
è completato per aggiunta
di TFIIE e TFIIH.
ZATE
S II CTD della RNA
polimerasi è sottoposto
a fosforilazione.
P
P
P
P
Inizia la trascrizione
- Legame sequenziale di sei fattori di
trascrizione generali e della RNA
polimerasi. - L'attivazione della RNA polimerasi
richiede una fosforilazione ATP-
dipendente. - Intervento di proteine aggiuntive
che despiralizzano la molecola di
DNA facilitando l'assemblaggio del
complesso a livello di specifici geni.