Genetica Molecolare: trascrizione e traduzione del DNA, lezione 9
Documento sulla Genetica Molecolare - Lezione 9 - Prof. Federico Gulluni. Il Pdf, utile per studenti universitari di Biologia, esplora la trascrizione e traduzione del DNA, il codice genetico, il controllo idrolitico e la struttura dei ribosomi.
Mostra di più17 pagine

Visualizza gratis il Pdf completo
Registrati per accedere all’intero documento e trasformarlo con l’AI.
Anteprima
GENETICA MOLECOLARE
INTRODUZIONE ALLA LEZIONE
Le scorse lezioni hanno riguardato l'informazione genetica, ovvero quella contenuta all'interno dei geni. Questi, trasmissibili alle cellule figlie, possono essere espressi formando un trascritto che, al termine della replicazione, viene tradotto in proteine.
RIASSUNTO GENERALE
Tramite quest'immagine riassuntiva è possibile visionare gli argomenti affrontati nelle scorse lezioni, ovvero le Polimerasi I-II-III, responsabili della trascrizione di RNA diversi che cooperano al livello del processo traduzionale.
- Dalla Pol II (lato sinistro dello schema) vediamo l'RNA più semplice, ovvero I'RNA messaggero, che viene modificato con il CAP al 5', la coda di poli A all'estremità 3' e lo splicing delle sequenze introniche. Viene successivamente portato fuori dal nucleo, raggiungendo il citoplasma;
- Dalla Pol III (al centro) otteniamo il tRNA che ha una struttura differente rispetto all' mRNA. Anche lui viene esportato dal nucleo al citoplasma e subisce degli editing (processi di modificazione). I più importanti servono per permettergli di legare gli amminoacidi che servono a ricopiare l'informazione presente nel codone;
· La Pol I (lato destro) trascrive l'rRNA (RNA ribosomiale) il quale si assembla già a livello del nucleolo. Il complesso ribosomiale, formato da subunità maggiore e minore, esce già formato dal nucleo e andrà a partecipare al processo di traduzione. Nella parte inferiore dell'immagine invece viene mostrato il processo post-trascrizionale dell'espressione genica, che permette il passaggio allo step successivo: la traduzione.
LA TRADUZIONE
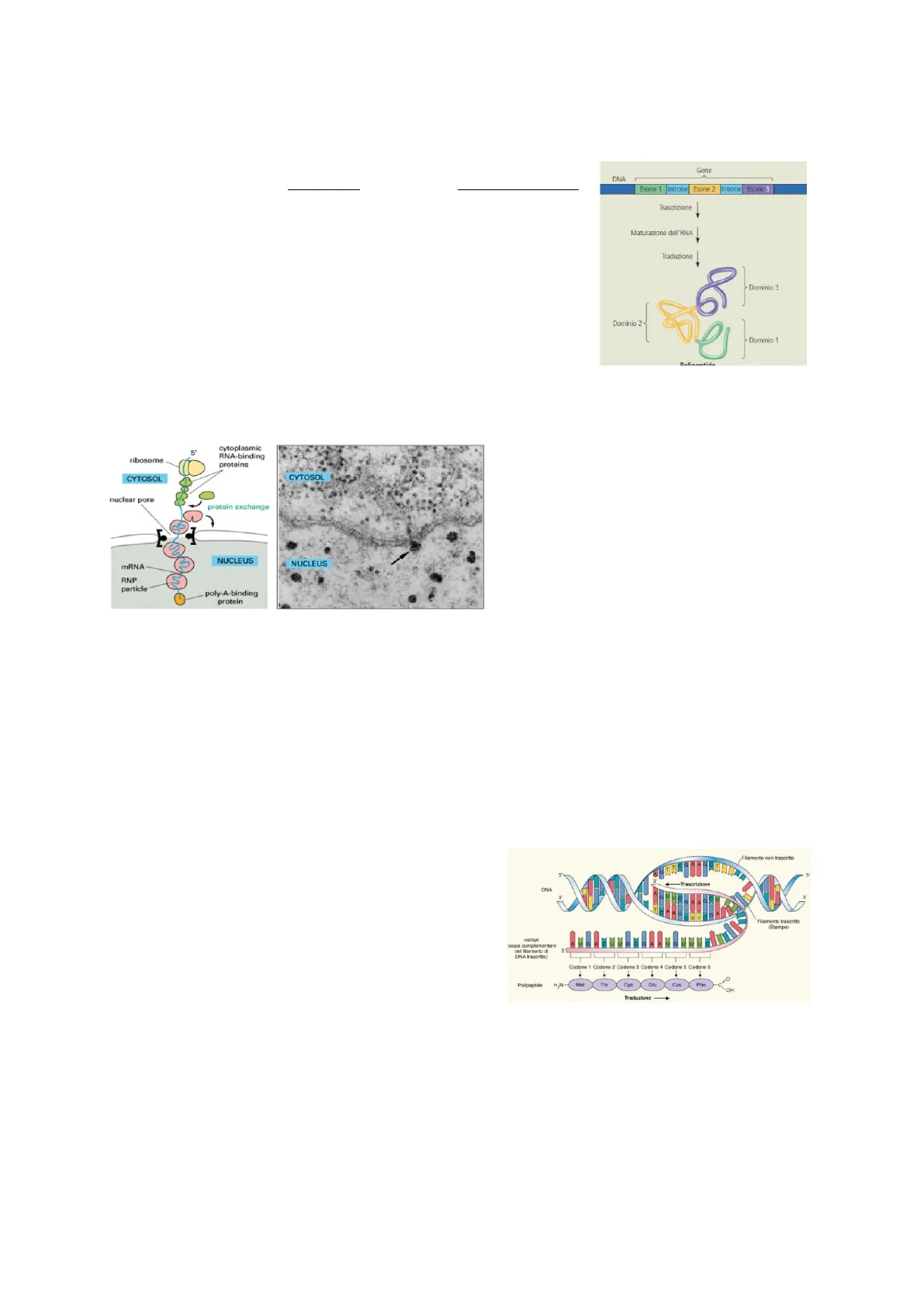
Come detto nella lezione precedente, le sequenze geniche contengono sequenze codificanti (esoni) e non condificanti (introni). Nel processo di traduzione vengono espresse le regioni esoniche. Solitamente, ogni regione esonica corrisponde ad un determinato dominio funzionale della proteina (nella ricerca, vengono solitamente isolati i vari esoni per studiare singolarmente i singoli domini). Come si vede nell'immagine, la sequenza singola dell'esone 1 verde darà origine ad un polipeptide verde chiamato dominio 1 e la regione dell'esone 2 origina il dominio 2, così come l'esone 3 forma il dominio 3. Insieme, danno una proteina funzionale multidominio.
Per portare I'mRNA fuori dal nucleo, attraverso i pori nucleari, sono necessarie delle proteine dette ribonucleoproteine (piccole molecole di RNA). Queste servono a rivestire e a mascherare la carica dell'mRNA, data dallo scheletro di gruppi fosfati. Queste proteine (visibili in rosa nell'immagine), una volta giunte nel citoplasma, si staccano e rientrano nel nucleo tramite un processo di importazione nucleare. L'immagine a destra mostra, tramite microscopia elettronica, le stesse proteine presenti a sinistra: la doppia linea presente nel mezzo corrisponde alla doppia membrana nucleare, con sopra la regione citosolica e sotto quella nucleare. I pallini neri più piccoli rappresentano i ribosomi che traducono I'mRNA uscente ed i pallini più grandi nella regione nucleica sono sequenze di mRNA rivestite dalle ribonucleoproteine che si assottigliano per passare nei pori.
IL CODICE GENETICO
Quando I'mRNA raggiunge il citoplasma, deve essere decodificato in modo da tradurre la sequenza nucleotidica in una sequenza amminoacidica. Occorre però notare che mentre i nucleotidi sono 4, gli amminoacidi sono 20. Vista l'incongruenza numerica, ipotizzare che un singolo nucleotide corrisponde ad un singolo amminoacido non è corretto. Ipotizzando sequenze di due nucleotidi per ogni amminoacido si giunge ad un massimo di 16 amminoacidi differenti, quindi troppi pochi rispettoa al numero reale: anche questa ipotesi è errata. La soluzione che si è rivelata corretta è l'utilizzo di un codice a triplette: usando una sequenza di 3 nucleotidi e leggendo l'mRNA in direzione 5'-3', è possibile non soltanto specificare tutti i 20 amminoacidi, ma anche avere più triplette in grado di codificare per uno stesso amminoacido.
DECODIFICARE IL CODICE
La scoperta del codice a triplette deriva da tre ricercatori: Nirenberg, Holley e Khorana, che nel 1968 ricevettero il Nobel per la definizione del codice genetico. Sono stati fatti degli studi per capire effettivamente quale fosse l'amminoacido corrispondente ad una determinata tripletta, partendo con un esperimento semplice, come quello di Nirenberg e Matthaei.
I due ricercatori hanno preso una serie di nucleotidi tutti uguali (in questo caso tutti di uracile) e hanno creato un polimero di poli-U, un tipo di polipeptide formato dallo stesso amminoacido ripetuto molte volte. Hanno inserito il poli-U in delle provette contenenti tutto l'apparato traduzionale della cellula, inclusi i ribosomi. Erano presenti 20 amminoacidi, di cui 19 non marcati e uno marcato radioattivamente. Hanno lasciato che questo polimero venisse tradotto e hanno osservato quale provetta contenesse l'amminoacido radioattivo. In questo caso, la sequenza UUU corrispondeva all'amminoacido fenilalanina. Hanno poi fatto la stessa cosa con la citosina, con l'adenina e anche con la guanina, senza però riuscirci perché i poli G erano instabili.
Hanno analizzato poi una situazione più complessa (dato che non tutti gli AA sono formati dallo stesso nucleotide): sono andati a sintetizzare polimeri molto corti con una reazione in grado di aggiungere solamente tre nucleotidi, corrispondenti alle triplette. Incubavano queste triplette con degli tRNA, ciascuno legato con il proprio AA e poi con il ribosoma. A questo punto, essendoci una sola tripletta, si formava un complesso stabile nel quale il tRNA era appaiato col suo anticodone al codone e il complesso ribosomiale. Questo aveva un peso molecolare abbastanza grande da permettere la separazione del complesso da tutti gli altri tRNA. Quindi facevano passare questa miscela in una beuta nella quale era presente un filtro, in modo da far passare tutti i tRNA non legati, tranne quello con ingombro sterico maggiore, ovvero l'unico che era legato al suo amminoacido. Da quello determinavano l'aminoacido corrispondente e, con molta pazienza, sono andati a definire tutti gli altri.
Domanda: Con l'uso dei tRNA quali altri accoppiamenti fra codoni e amminoacidi potrebbero verificarsi?
Domanda: Quali amminoacidi vengono specificati da codoni composti da un solo tipo di base?
CORNICE DI LETTURA
Nel processo di traduzione, è importantissimo mantenere la cornice di lettura: una sua modifica può cambiare completamente gli amminoacidi (AA) specificati. È importante ricordare che il primo AA tradotto sarà sempre la metionina. Come mostrato nell'immagine, una mutazione può portare a un cambiamento nell'intera sequenza di AA.
IL CODICE A TRIPLETTE
L'immagine mostra l'intero codice genetico, costituito da 64 triplette. Il codice è: -universale: tutti gli organismi (batteri, eucarioti, ecc.) sono in grado di tradurre la stessa sequenza di mRNA nella stessa struttura proteica. -ridondante: un singolo amminoacido può essere specificato da più triplette che, nella maggior parte dei casi, si differenziano per il nucleotide nella terza posizione. Le uniche eccezioni sono la metionina (codone di inizio AUG) e il triptofano (UGG), entrambi specificati da una singola sequenza. Gli altri amminoacidi sono specificati da almeno due sequenze diverse.
È inoltre necessario sapere dove termina la traduzione, per questo esistono tre sequenze di stop (UAA, UGA, UAG). Nella parte inferiore dell'immagine sono presenti alcuni amminoacidi molto importanti a livello proteico, specificati da molti codoni (es. cisteina che serve per formare i ponti disolfuro). Questo è stato possibile grazie all'evoluzione, che ha permesso di contrastare le possibili mutazioni. -non ambiguo: una certa tripletta sarà sempre relativa ad un solo AA.
IPOTESI VACILLAMENTO III BASE
Ciò che accomuna la ridondanza dello stesso AA è che cambia quasi sempre il nucleotide in terza posizione . Prendiamo come esempio la prolina che ha una sequenza CC in posizione 1 e 2 e nella terza può essere una C, G, U oppure una A. In tutti e 4 i casi otterremo sempre una prolina. Ciò è stato teorizzato già nel 1966 da Crick ed è stata definita l'ipotesi del vacillamento della terza base. Questo è possibile perché il tRNA ha una struttura ad anse che presenta un anticodone che riesce a riconoscere con alta affinità la base 1 e 2, mentre l'amminoacido in posizione 3, trovandosi in una regione incurvata, può avere un appaiamento non proprio specifico.