Regolazione della trascrizione e splicing in genetica molecolare
Documento di Genetica Molecolare - Lezione 8 - Prof. Federico Gulluni sulla regolazione della trascrizione e i meccanismi di splicing. Il Pdf esplora introni, esoni, poliadenilazione e spliceosoma, contribuendo alla comprensione della complessità del proteoma. Questi appunti universitari di Biologia sono utili per lo studio autonomo.
Mostra di più13 pagine

Visualizza gratis il Pdf completo
Registrati per accedere all’intero documento e trasformarlo con l’AI.
Anteprima
GENETICA MOLECOLARE - Lezione 8 - Prof. Federico Gulluni
30/10/2024
SBOBINATORE: Costantini Antonio
REVISORE: Gatto Cristiano
LEZIONE 8 - BASI BIOLOGICHE, MOLECOLARI E DI GENETICA UMANA: MODULO DI GENETICA MOLECOLARE
INTRODUZIONE ALLA REGOLAZIONE DELLA TRASCRIZIONE
Nella scorsa lezione si è parlato della trascrizione e dei complessi relativi all'attivazione dell'RNA polimerasi.
La seconda lezione sarà dedicata principalmente ai meccanismi di regolazione della trascrizione e in modo
più specifico si vedrà come vi siano delle differenti espressioni a livello trascrizionale mediate da fattori di
regolazione selettivi.
PERCHÉ È IMPORTANTE LA REGOLAZIONE DELLA TRASCRIZIONE?
Partendo da questo esempio, si possono vedere
quattro organismi molto differenti l'uno dall'altro.
Si va da uno a minor complessità nel caso
dell'Escherichia Coli a uno a maggior complessità
come l'Homo sapiens. Se si guarda il contenuto a
livello di numero di geni di questi organismi si hanno all'incirca 4.000 geni nei batteri, 6.000 in una cellula
eucariote semplice come il lievito, 16.000 in un insetto, che è già un organismo più complesso rispetto ad
organismi unicellulari come il lievito o l'E.Coli, se si considera l'uomo si può vedere che il numero di geni
aumenta ulteriormente fino a circa 20.000/25.000 geni. Però si ha un numero di geni non così elevato
rispetto ad un organismo relativamente semplice come un insetto. Come è possibile che con solo 4.000 geni
in più si riesca a specificare un organismo tanto più
complesso? Il motivo è che in realtà non esiste una
relazione lineare tra il contenuto in termini di geni e il
contenuto in termini di proteine. Esiste, quindi, una
curva di complessità del proteoma (l'insieme di tutte le
proteine che si trova all'interno delle nostre cellule):
nonostante si parta da un numero fisso di geni, in
seguito alla trascrizione di quest'ultimi, il trascritto
generato va incontro a due eventi chiave:
- modificazioni post-trascrizionali,
- eventi post-traduzionali.
Tramite questi due eventi si riesce a modificare l'informazione contenuta originariamente nel gene e a
produrre, a partire da uno di essi, tipi di proteine differenti. Ad esempio, andando a rimuovere alcune
sequenze esoniche oppure tagliando a livello post-traduzionale dei pezzi di una proteina sintetizzata e
generare una differente, che avrà una funzione diversa. Quindi partendo da un numero di geni limitato
20/25.000, si arriverà a più di un milione di proteine.
INTRONI ED ESONI
Generazione di proteine multiple dallo stesso trascritto
A cosa è dovuta questa possibilità di generare proteine multiple e partire dallo stesso trascritto?
Nei geni degli organismi più complessi, la sequenza di un gene non è formata esclusivamente da regioni
codificanti. Si ha una gran quantità di sequenze non codificanti.
I geni eucariotici contengono porzioni
codificanti (esoni, in rosso nell'immagine)
e porzioni non codificanti (introni, in giallo
nell'immagine). L'mRNA corrispondente
contiene sia esoni che introni.
In un modello semplificato si rimuovono le regioni introniche e si attaccano le regioni esoniche l'una vicino
all'altra producendo mRNA codificante. Inoltre, andando a modificare la composizione esonica, si può
alterare la sequenza di una proteina e quindi la sua struttura. Questo permette di capire come basti un
numero di geni ridotto, per essere degli organismi più complessi rispetto ad un moscerino.
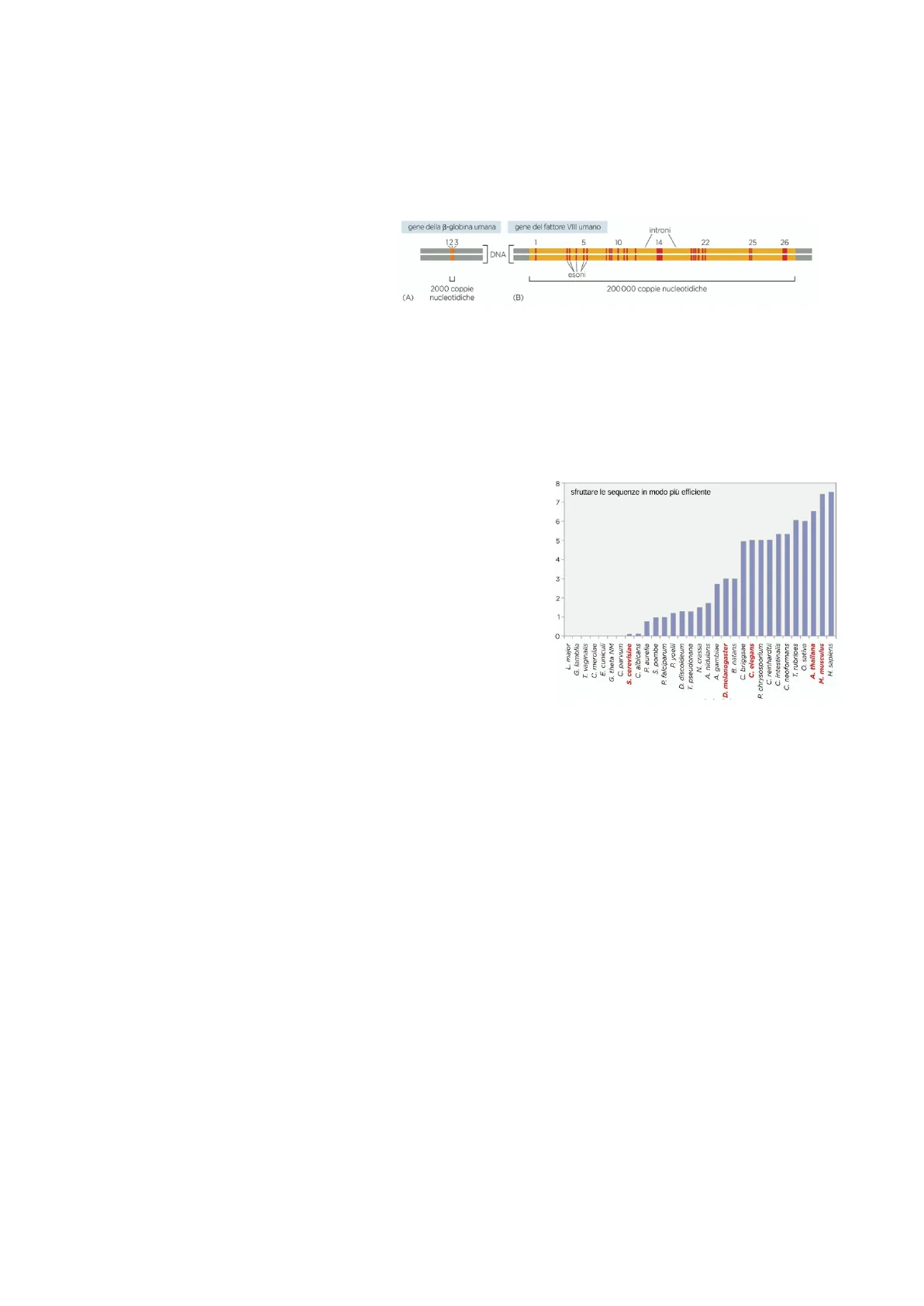
Numero di introni e complessità degli organismi
Facendo riferimento all'immagine a destra si nota che il numero
di introni, quindi regioni non codificanti, ha un andamento
lineare che tende ad aumentare all'aumentare della complessità
degli organismi. Infatti i batteri non hanno assolutamente introni;
quindi, nella maggior parte dei casi il loro RNA messaggero verrà
tradotto così com'è in una proteina (da 4.000 geni si ottengono
circa 4.000 proteine). Il lievito ha un numero di introni per gene
bassissimo: sotto 1. Quindi il lievito, pur essendo un organismo
eucariote, è molto semplice e non ha bisogno di una regolazione
dell'espressione genica perché è un organismo unicellulare che
ha bisogno di produrre le proteine che gli servono per dividersi. Passando alla Drosophila, presenta un
numero intermedio di introni (circa 3 introni per gene).
Differenza tra lievito e Drosophila
Qual è dunque la differenza tra il lievito e la Drosophila?
Il primo è un organismo unicellulare e il secondo è pluricellulare. Questa è la prova del fatto che più un
organismo è complesso più c'è bisogno di regolare in maniera più fine l'espressione genica.
L'Homo sapiens, ha una media di 7/8 sequenze introniche per gene. Man mano che gli organismi sono
diventati più complessi è aumentata anche la quantità di DNA intronico, non solo in termini di lunghezza,
ma anche in termini di numero.
Quindi si può dire che in realtà quello che ha permesso di diventare degli organismi più complessi, non è
tanto la distinzione in nuove sequenze geniche, quanto la possibilità di sfruttare quelle determinate
sequenze geniche che si avevano in maniera più efficiente, producendo varianti che hanno assunto delle
funzionalità diverse all'interno della cellula.
Il professore accenna un'esperienza personale: "Molti anni fa ho iniziato a fare ricerca studiando un gene
della Drosophila, che esisteva in una singola coppia genica. Questo gene esiste in tre coppie dell'uomo,
quindi, è aumentato a livello di contenuto in termini di geni e di duplicazione, ma poi esistono nell'uomo
degli eventi di splicing, come vedremo, che portano alla rimozione di determinate sequenze, le quali
permettono di ottenere un numero di proteine più alto rispetto a quel gene che avevamo nella Drosophila."
I TRE EVENTI POST-TRASCRIZIONALI
Variazione a livello del trascritto
Come si riesce a definire una variazione a livello del trascritto?
I tre elementi principali per la modifica del mRNA sono:
- Capping, cioè l'aggiunta di un cappello protettivo per I'mRNA in posizione 5', formato da
7-metilguanosina, e serve per evitare la degradazione del trascritto da parte delle nucleasi, - Poliadenilazione, serve per dare stabilità al trascritto e per essere esportato dal nucleo,
- Splicing, rimozione delle sequenze introniche. In alcuni casi viene definito splicing alternativo
perché permette di rimuovere anche le sequenze esoniche, generando un prodotto proteico
differente.
Nei procarioti tutto ciò non avviene.
CAPPING
Aggiunta del cappuccio protettivo
All'estremità 5' del trascritto, si trova il primo nucleotide aggiunto
dalla RNA polimerasi, il quale presenta tre gruppi fosfato. Alcuni
enzimi specifici sono in grado di aggiungere al primo nucleotide un
cappuccio formato da 7-metilguanosina (con una struttura ribaltata
rispetto al normale andamento dei nucleotidi, come in foto) che si
lega al mRNA con un legame 5'-5'. LA 7-metilguanosina non viene
aggiunta nel momento in cui viene sintetizzato il primo nucleotide,
ma deve essere raggiunta una lunghezza del trascritto di circa 20/30
nucleotidi, a causa dell'ingombro sterico, per non interferire con il
complesso del RNA polimerasi. La 7-metilguanosina conferisce
stabilità e protezione al trascritto.
Sequenza di eventi per la formazione della 7-metilguanosina
In quest'immagine si vede la sequenza di eventi con cui si
forma la 7-metilguanosina. All'inizio c'è un trascritto di mRNA
con tre gruppi fosfato all'estremità 5'. Ma per formare un
legame è necessario rimuoverne uno. Grazie ad un RNA
trifosfatasi, cioè un enzima che riconosce i nucleotidi
trifosforilasi, è possibile fare quanto detto. Avendo solo due
fosfati legati al primo nucleotide, può arrivare un enzima
chiamato guanil-transferasi, il, quale aggiunge un nucleotide di
guanina con il suo gruppo fosfato, catalizzando un legame 5'-5'.
Segue poi una metil-transferasi (un enzima in grado di
attaccare i gruppi metile) che aggiunge un gruppo metile in
posizione 7 della guanina. Questo tipo di enzima può
aggiungere altri gruppi metilici anche in posizione diverse,
come nel primo, secondo e terzo nucleotide. Questi gruppi
metilici impediscono che il trascritto venga degradato prima di
essere tradotto in proteina.
La 7-metilguanosina viene aggiunta solo agli RNA messaggeri e non a quelli di trasporto o ribosomiali.
Questo perché la guanil-transferasi è associata alla RNA polimerasi II, che trascrive i pre-mRNA, ma è
assente nella RNA polimerasi I e III, che trascrivono l'rRNA e i tRNA.
POLIADENILAZIONE
Regioni del trascritto e loro funzione
Come si può vedere dall'immagine nel
trascritto
ci
sono delle regioni
codificanti
e non codificanti, in
particolar modo prima del codone
d'inizio avremo una regione 5' UTR,
(UnTranslated Region) una regione che non verrà tradotta. Poi c'è un'altra regione non tradotta 3' UTR in
fondo al trascritto. Queste due regioni non portano ad un prodotto proteico, ma servono a regolare
l'esportazione e la traduzione dell'mRNA. La sequenza codificante occupa una regione più piccola, centrale.
La poliadenilazione viene aggiunta al termine della posizione del trascritto, consiste in una coda di adenine,
di lunghezza variabile (ca. da 50 fino a 250/300 adenine). Le adenine servono per conferire maggiore o
minore stabilità nel trascritto. C'è una relazione diretta tra la quantità di RNA messaggero e la quantità di
proteine, se si modula la stabilità dell'RNA messaggero, aggiungendo più o meno adenine, si modula
anche il tempo impiegato nella traduzione in proteina, di conseguenza si andrà controllare la quantità di
proteina prodotta per l'unità di RNA messaggero.
Processo di aggiunta della coda di poli-A
Per aggiungere la coda di poli-A, nel momento in cui la
trascrizione arriva verso la fine, esiste una sequenza
consenso (sequenza di nucleotidi conservata, AAUAAA
nell'immagine), che viene riconosciuta da alcuni enzimi, i
quali segnalano che la trascrizione è quasi verso la fine; a
questo punto avviene un taglio, dopo circa 11-30
nucleotidi, grazie a dei fattori di taglio, i quali
rimuoveranno l'RNA messaggero.
Meccanismo di poliadenilazione
Come si può notare dall'immagine, al termine della trascrizione
c'è questa sequenza che segnala la fine del trascritto, essa viene
legata al fattore trascrizionale CPSF (fattore di stimolazione del
taglio), che recluta i fattori di specificità del taglio: CSTF.
Quest'ultimo a sua volta recluta le proteine CFI (fattore di taglio
1) e CFII (fattore di taglio 2) (in verde) che incurvano
l'RNA messaggero, essi sono in grado di tagliare questa
porzione incurvata, e di conseguenza porre fine alla lunghezza
del trascritto. Solo a questo punto, quando è avvenuto il taglio,
le proteine di stimolazione del poli-a reclutano le poli-a, a livello
della regione terminale, dove verrà aggiunta la coda di
poliadenilazione. A questo punto il trascritto può essere
trasportato a livello citoplasmatico.
SPLICING
Rimozione delle porzioni introniche
Il terzo evento importante di regolazione che avviene durante il processo di
trascrizione, è lo splicing. Quest'evento consiste nella rimozione delle porzioni
introniche (abbastanza ampie e variabili in lunghezza), a seguito di un
ripiegamento di queste ultime. Queste zone vengono definite interposte tra
sequenze esoniche, le quali verranno espresse.