Espressione genica: trascrizione del DNA in RNA e maturazione dell'mRNA
Documento sull'espressione genica, trascrizione del DNA in RNA e maturazione dell'mRNA. Il Pdf esplora le differenze tra procarioti ed eucarioti, la struttura dell'RNA polimerasi e le modifiche post-trascrizionali, utile per studenti universitari di Biologia.
Mostra di più37 pagine

Visualizza gratis il Pdf completo
Registrati per accedere all’intero documento e trasformarlo con l’AI.
Anteprima
Espressione Genica
L'espressione genica è il processo mediante il quale l'informazione contenuta nel gene viene convertita in una macromolecola funzionante, la proteina.
Trascrizione
Un passaggio cruciale nell'espressione genica è la copiatura del filamento stampo di un gene in un trascritto complementare di RNA (mRNA). Dunque la trascrizione è quel processo intermedio tra la replicazione e la traduzione che ci permette di trasformare l'informazione contenuta nel DNA in intermedio strutturale del RNA. Per parlare di trascrizione è opportuno conoscere la corretta terminologia; del doppio filamento di DNA:
- Filamento codificante > portatore dell'informazione, la cui sequenza è identica a quella dell'RNA trascritto; ovviamente questo filamento sarà complementare al filamento stampo
- Filamento stampo > su questo filamento avviene il processo di polimerizzazione e complementare alla sequenza di RNA
Unità Trascrizionale
UNITA' TRASCRIZIONALE = è identificata da un punto d'inizio (dove inizia la trascrizione) e un punto di fine (terminatore). Questa regione per convenzione è numerata progressivamente in positivo a partire dal primo nucleotide trascritto, a cui viene attribuita come numerazione +1 fino all'ultimo nucleotide del trascritto. Le regioni del DNA che si trovano a monte del punto d'inizio della trascrizione verranno indicate con una numerazione negativa: - 1 è il primo nucleotide che si trova a monte del punto d'inizio della trascrizione. Da questa regione spostandoci in direzione 3' -- >5', tutti gli altri nucleotidi avranno numerazione negativa crescente.
Maturazione negli Eucarioti
Negli eucarioti è importante la fase di maturazione: splicing; ciò poiché gli eucarioti presentano delle sequenze non codificanti di regolazione della trascrizione fondamentali nell'avere un prodotto nella traduzione che sia aderente poi all'informazione che era presente nel DNA. Queste sequenze non codificanti si chiamano introni e dovranno essere eliminate in modo tale da avere solamente l'insieme di geni (o operoni) codificanti chiamati ORF che poi saranno il substrato per la traduzione.
Trascrizione e Traduzione nei Procarioti
I procarioti non presentano queste sequenze non codificanti. La particolarità dei procarioti è anche il fatto che non hanno il nucleo, ma nucleoidi che sono delle aree deputate alla localizzazione del materiale genico nelle cellule procariote dove non c'è una distinzione netta tra plasma nucleare e organello. Nei procarioti trascrizione e traduzione sono due processi combinati perché appena il filamento singolo di RNA neo-formato viene allungato fuori dalla bolla di replicazione del DNA e incontra il primo ribosoma avviene direttamente la traduzione. Dunque, la trascrizione inizia e avviene in punti dei nucleoidi dove c'è maggiore addensamento ribosomiale, ciò poiché si avrà maggiore possibilità che il filamento neoformato sia tradotto.Non appena la trascrizione ha inizio e l'estremità 5' dell'mRNA è accessibile, un ribosoma vi si attacca e inizia la sintesi proteica, consentendo al processo combinato di trascrizione e traduzione di terminare in pochi minuti. Una volta che il primo ribosoma ha lasciato l'estremità 5', un secondo ribosoma si può attaccare.
Trascrizione e Traduzione nelle Cellule Eucariote
Viceversa, le cellule eucariote presentando il nucleo si avrà che la trascrizione avverrà nel nucleo, successivamente avviene lo splicing per avere un filamento di mRNA maturo, questo poi fuoriuscirà dal nucleo e passerà nel citoplasma per trasporto attivo, dove avverrà la traduzione. La trascrizione trasforma l'informazione contenuta nella sequenza di basi del DNA in una sequenza nucleotidica di RNA. La sintesi dell'RNA è catalizzata dalla RNA polimerasi.
RNA Polimerasi
L'RNA polimerasi è un enzima che non ha bisogno di energia scaturita dall'utilizzo dell'ATP, non ha bisogno di innesco in quanto può iniziare la trascrizione ex novo (diversamente la DNA polimerasi). Le RNA polimerasi sintetizzano RNA in direzione 5'->3' utilizzando i 4 tipi di riboNTP come precursori e copiando lo stampo di DNA.
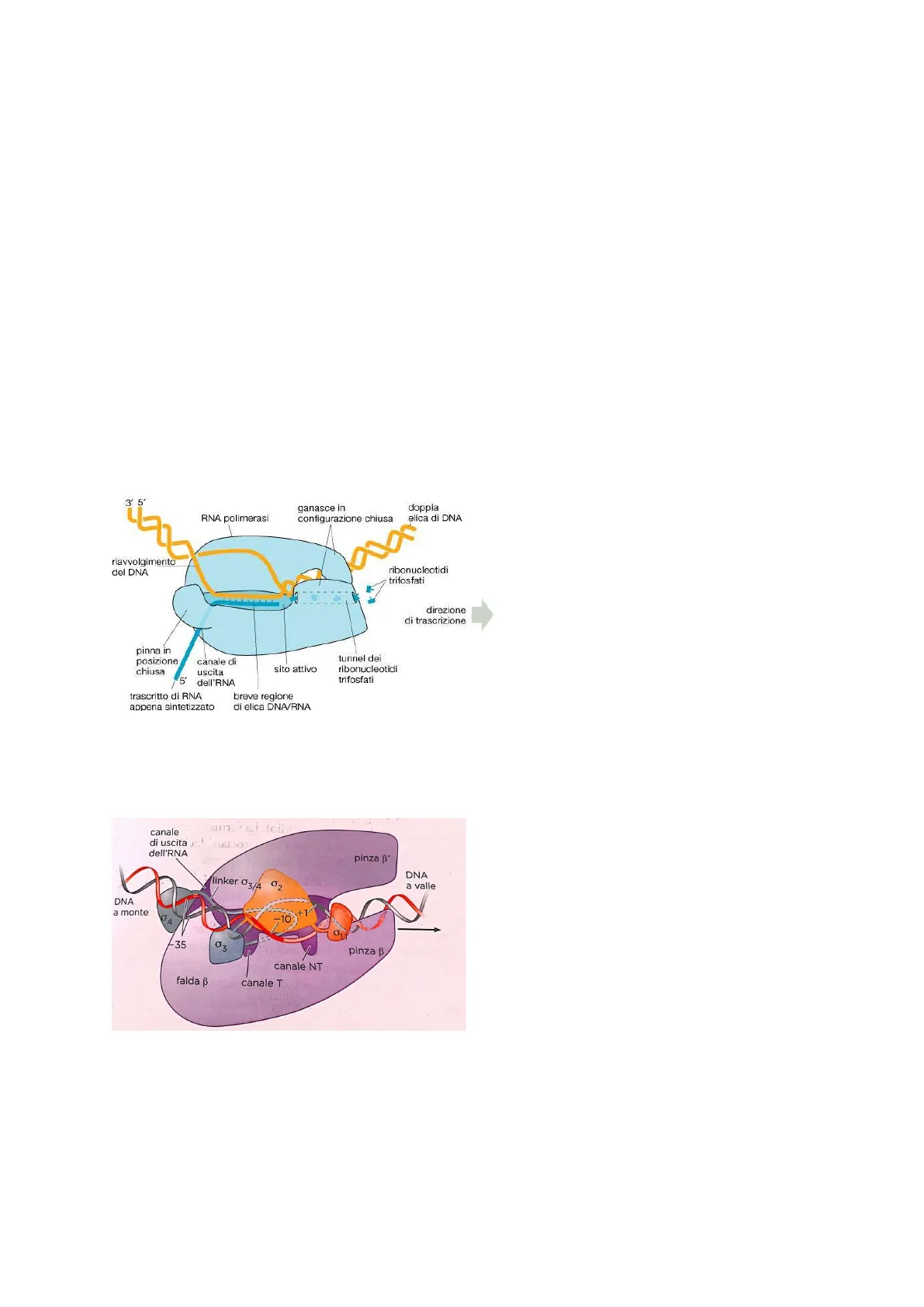
3' 5' ganasce in configurazione chiusa L'RNA polimerasi viene rappresentata RNA polimerasi doppia elica di DNA strutturalmente come la chela di un granchio: RNA polimerasi si avvolge riavvolgimento del DNA ribonucleotidi trifosfati attorno al DNA agganciandosi con due parti come fossero appunto le due parti direzione di trascrizione costituenti della chela di un granchio. pinna in posizione canale di uscita sito attivo 5' dell'RNA tunnel dei ribonucleotidi trifosfati chiusa Il movimento dell'enzima si può immaginare in questo modo: la chela che trascritto di RNA appena sintetizzato breve regione di elica DNA/RNA scorre sul DNA oppure viceversa il DNA scorre attraverso la chela formando la bolla di trascrizione. Sulla struttura dell'RNA polimerasi abbiamo un punto di svolgimento e un punto di riavvolgimento del DNA e al centro di questa regione si ha il sito catalitico. In corrispondenza del sito catalitico, vi è un punto in cui l'RNA nascente fuoriesce attraverso un canale d'uscita.
canale di uscita dell'RNA Le due componenti della chela (subunità beta pinza B' e beta 1) stabilizzano il DNA in entrata e poi K linker 03/4 º2 DNA a valle DNA a monte avremmo un canale centrale (NTP-uptake) che permette l'entrata dei riboNTP , questi 04 03 canale NT verranno avvicinati per la formazione del -35 -10 legame fosfodiesterico con conseguente pinza ₿ uscita del pirofosfato. Nel sito attivo non ci falda ß canale T saranno due magnesi ma uno solo che contribuisce alla catalisi fosfodiesterica. All'interno del canale centrale i filamenti di DNA si allontanano dalla posizione + 3. Il filamento che non fa da stampo esce attraverso il canale NT (nontemplate) e si sposta lungo la superficie dell'enzima. Il filamento stampo, invece, segue un percorso lungo il canale centrale ed esce attraverso il canale T (template). La doppia elica si riforma in posizione - 11 nel DNA a monte, cioè dietro l'enzima. Poi c'è la componente del canale centrale che permette la fuoriuscita del RNA neo-formato. RNA neo-formato e DNA stanno tra loro a 90°.
RNA Polimerasi Batterica
RNA polimerasi batterica è costituita da 6 subunità (5 core + sigma) che sono:
- 2 subunità alfa (40kd)
- subunità beta
- subunità beta 1
- subunità omega
- fattore sigma
Queste formano l'oloenzima che ha un peso di circa 465 kd . La subunità catalitica principale quella delle 2 chele sono le subunità beta e beta 1 che sono le subunità che stabilizzano la molecola del DNA all'interno dell'enzima mentre il fattore sigma serve per posizionare l'RNA polimerasi (le subunità beta e beta 1) alla molecola del DNA. Il gene RPOC codifica le subunità beta1: una sua mutazione porterebbe all'incompatibilità con la vita.
Fattore Sigma
Il fattore sigma (sigma70) è codificato dal gene RPOD. Sigma 70 conferisce all'RNA polimerasi la specificità per il punto di inizio della trascrizione, ossia la capacità di riconoscere il promotore. L'N-terminale di sigma controlla l'attacco al DNA L'N-terminale blocca l'attacco al DNA nell'oloenzima Domini di legame al DNA Regione N-terminale Il DNA sposta I'N-terminale nel complesso aperto Regione N-terminale Questo riconoscimento deve avvenire solo quando la proteina è associata al core enzimatico dell'RNA polimerasi. Per cui la struttura della proteina è tale da prevenire la capacità del fattore sigma di legare il promotore in assenza del core enzimatico: ciò è dovuto al fatto che la regione N-terminale nelle condizioni in cui sigma70 è libero, non associato all'enzima, è ripiegata a coprire le eliche del C- terminale che prendono contatto con il DNA. Soltanto quando si forma il complesso, la regione N-terminale cambia posizione e libera, espone le alfa eliche che prenderanno contatto con il promotore.
Velocità e Superavvolgimenti
La velocità dell'RNA polimerasi batterica è 40 nt/sec, nei lieviti (fagi) è 200 nt/sec. Inoltre, RNA polimerasi avrà il compito di svolgere i superavvolgimenti del DNA: avremmo un superavvolgimento negativo che per convezione lo indicheremmo come in uscita dopo la trascrizione mentre quello positivo in entrata
Tipi di RNA Polimerasi
I procarioti hanno una sola RNA polimerasi, mentre gli eucarioti ne hanno tre: le RNA polimerasi I, Il e III. La composizione in basi della catena di RNA neosintetizzato è complementare a quella della catena stampo di DNA (filamento antisenso), ed esattamente simile a quella della catena codificante di DNA (filamento senso), fatta eccezione per la presenza dell'uracile al posto della timina. Per trascrivere un gene, la RNA polimerasi procede attraverso una serie di passaggi ben definiti suddivisi in tre fasi: inizio, allungamento e terminazione.
Fasi della Trascrizione
1. Inizio della Trascrizione
1. INIZIO La trascrizione inizia a livello di determinate regioni del DNA poste al 5' del gene. Tali regioni costituiscono il promotore del gene. Dunque possiamo dire che la trascrizione ha inizio con il legame dell'RNA polimerasi al promotore; questo legame forma il "complesso chiuso". In questa forma il DNA rimane a doppia elica e l'RNA polimerasi sarà legato ad una faccia dell'elica. Nel secondo passaggio dell'inizio, il complesso chiuso subisce una transizione verso un complesso aperto, in cui le catene di DNA si separano per ~ 13 coppie di basi attorno al punto di inizio formando la "bolla di trascrizione". Ricorda: denaturazione non necessità di ATP, è il risultato di un cambiamento conformazionale del complesso DNA-enzima verso una forma energeticamente più favorevole. Questa apertura (melting) avviene tra le posizioni -11 +2. Nella successiva fase d'inizio, l'RNA polimerasi comincia la sintesi di RNA per poi rilasciare il promotore. Una volta che l'enzima ha superato i primi 10 nucleotidi si dice che è evaso dal promotore. A questo punto si è formato un complesso ternario stabile, contenente l'enzima, il DNA e RNA. Questa è la transizione verso la fase di allungamento.
Promotore e Sequenze Consenso
Il promotore è una sequenza conservata perchè presenta una sequenza nucleotidica ben precisa che è uguale in tutti i genomi.
- I promotori dei geni dei procarioti presentano due sequenze conservate a monte del sito di inizio della trascrizione. Queste due sequenze sono localizzate in una regione nota come "nucleo del promotore", in posizione ben precisa rispetto al sito di inizio della trascrizione (-10 e -35, separate dallo spaziatore ottimale della lunghezza di 17pb) Perché meno? Per convenzione, il meno indica tutte quelle sequenze che sono a monte della trascrizione. Il promotore è collocato in punti ben precisi che sono con un'alta frequenza di ripetizione all'interno delle sequenze conservate tanto da essere definite sequenze consenso che distano dal punto di inizio della trascrizione da -35 a -10, la -10 sarà quella più vicina all'inizio della trascrizione sino a -1 dove finisce il promotore e a +1 inizia la trascrizione.
+1= Sito di inizio PROMOTORE TERMINATORE Ex1 Int1 Ex2 Int2 Ex3 In particolare nei procarioti nella regione -10 c'è un abbondanza di TA che va a costituire una sequenza (TATAAT) chiamata TATA BOX che ha una minore forza per la presenza di 2 soli legami idrogeno, i quali necessitano di poca energia per l'apertura della bolla. Nella regione -35 ci sarà una sequenza TTGACA. Negli eucarioti non è sempre presente il TATA BOX, ci possono essere delle sequenze che hanno una maggiore ricchezza di GC.