Tecnologie del DNA ricombinante, espressione genica e clonaggio del DNA
Documento di Università su tecnologie del DNA ricombinante, espressione genica e clonaggio del DNA. Il Pdf, utile per lo studio della Biologia a livello universitario, approfondisce i sistemi di espressione, i vettori e i protocolli di clonaggio, con un focus sulla produzione di proteine ricombinanti.
Mostra di più42 pagine

Visualizza gratis il Pdf completo
Registrati per accedere all’intero documento e trasformarlo con l’AI.
Anteprima
Tecnologie Ricombinanti e Manipolazione del DNA
Le tecnologie ricombinanti sono l'insieme di procedure tecnologiche che, attraverso la manipolazione del DNA, consentono l'analisi di differenti prodotti genici ed eventualmente di regolarne l'espressione anche in contesti allotipici.
Conoscenze Necessarie per le Tecnologie Ricombinanti
È necessario conoscere:
- Le caratteristiche e la struttura dei geni e genomi -> La differenza si nota soprattutto nella grandezza delle regioni codificanti e non codificanti.
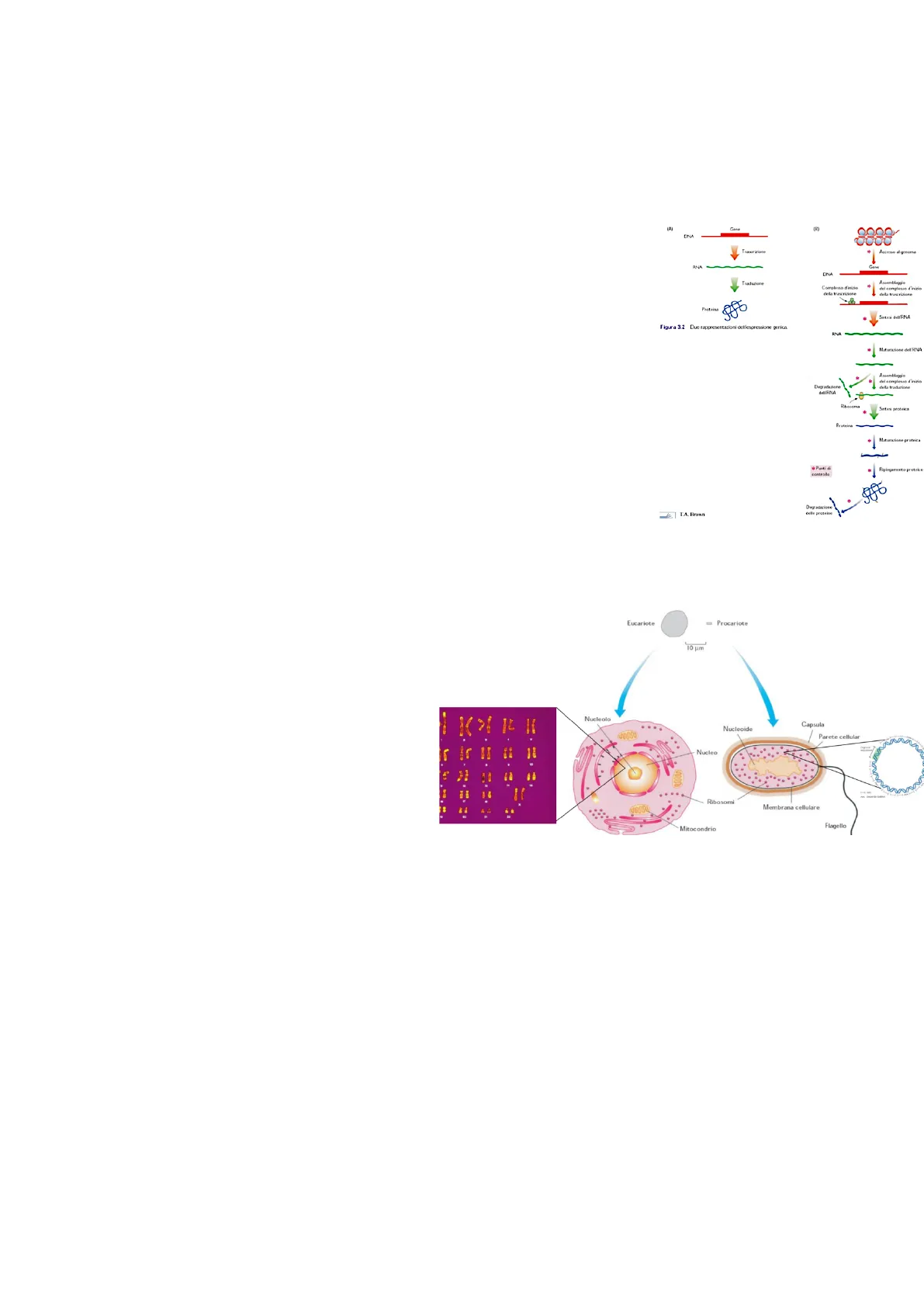
I meccanismi che regolano l'espressione genica -> In una cellula procariotica il gene viene trascritto in RNA e l'RNA viene subito tradotto in proteina (la traduzione avviene in contemporanea alla trascrizione per minimizzare la spesa energetica e perché non c'è compartimentalizzazione nella cellula procariotica). In una cellula eucariotica invece prima il DNA viene reso accessibile al macchinario trascrizionale, poi l'RNA viene trascritto in forma immatura per cui deve maturare, una volta che è maturo deve essere spostano nel citoplasma, dove viene letto dai ribosomi, i quali traducono l'RNA in proteina, poi la proteina verrà ripiegata per diventare funzionale.
(A) Gene (B) DNA Trascrizione Accesso al genoma RNA Gene DNA Assemblaggio Complesso d'inizio della trascrizione del complesso d'inizio della trascrizione Proteina Figura 3.2 Due rappresentazioni dell'espressione genica. ANA Maturazione dell'RNA Assemblaggio Degradazione del complesso d'inizio della traduzione dell'RNA Ribosoma Sintesi proteica Proteina Maturazione proteica · Punti & controllo . Ripiegamento protekce 1.80 Degradazione ! delle proteine T.A. Brown
Occorre considerare le caratteristiche strutturali e riproduttive degli organismi oggetto delle manipolazioni genetiche -> La riproduzione è diversa se si tratta di organismi unicellulari o organismi superiori (topi o uomo). Le cellule procariotiche non presentano un nucleo Eucariote = Procariote ma un nucleoide e non hanno una 10 um compartimentalizzazione interna, c'è una parete interna e una parete esterna. Le cellule eucariotiche hanno solo una membrana cellulare e un sistema di Nucleolo .. Nucleoide Capsula endomembrane che formano la tipica Nucleo 3 . compartimentalizzazione eucariotica eta (reticolo endoplasmatico, apparato di Golgi, mitocondri ecc). Entrambe le Ribosomi Membrana cellulare cellule hanno un citoplasma. Anche il Mitocondrio Flagello materiale genetico è diverso: nell'uomo abbiamo delle molecole di DNA organizzate in cromosomi contenuti nel nucleo (23 coppie di cromosomi di cui una sono cromosomi sessuali); il materiale genetico di una cellula procariotica invece è rappresentato da una molecola di DNA circolare, con elevata densità di sequenze codificanti. Parete cellular
Clonaggio di Geni e DNA Ricombinante
La più frequente applicazione tecnologica del DNA ricombinante riguarda l'espressione arbitraria di un gene. Per questo scopo, il gene deve essere clonato.
Il clonaggio di un gene consiste nell'identificare e isolare una sequenza di DNA di interesse, amplificare il frammento di interesse inserendolo in un vettore plasmidico, ossia un DNA usato come veicolo per trasferire geni, per creare una molecola ibrida di DNA ricombinante. Questa molecola ibrida viene trasferita in batteri affinché i batteri, moltiplicandosi, permettano di avere più copie del DNA iniziale selezionato. Generalmente si usano i batteri perché sono organismi semplici, economici e manipolabili, si riproducono in fretta quindi in breve tempo possiamo avere numerose molecole di DNA.
Traduzione Sintesi dell'RNAPer clonare il gene bisogna isolare il frammento di interesse e inserirlo in un vettore, che in genere è un DNA plasmidico, ossia una piccola molecola di DNA di origine procariotica che ha delle caratteristiche ben precise. L'inserzione del frammento di DNA nel vettore avviene tramite l'utilizzo di enzimi di restrizione. In questo modo si ottiene il vettore con all'interno il frammento di DNA d'interesse, questo prende il nome di DNA ricombinante. Per molecole di DNA ricombinante si intende una molecola di DNA costituita da frammenti di DNA di origine diversa. La molecola di DNA ricombinante deve essere prodotta in grande quantità, per cui viene inserita nei batteri. Successivamente bisogna selezionare solo i batteri che hanno internalizzato la molecole di DNA ricombinante. Poi i batteri vengono depositati su un terreno liquido, ossia una sostanza acquosa ricca di sostanze nutritive che permettono ai batteri di proliferare. I batteri proliferano e ogni volta che la cellula batterica replica, replica anche il vettore che abbiamo inserito. I batteri vengono lisati per poi isolare la grande quantità di DNA che abbiamo selezionato all'inizio.
Tecniche del DNA Ricombinante
Le tecnologie del DNA ricombinante si occupano di analizzare e modificare sequenze di DNA codificanti e non codificanti.
Con il termine di tecnologie del DNA ricombinante si intendono tutte quelle tecniche che permettono di:
- Isolare
- Purificare
- Analizzare
- Manipolare Le sequenze di DNA.
Per "DNA ricombinante" si intende il DNA proveniente da due o più origini diverse, combinati insieme (molecole ibride di DNA).
Queste tecniche vengono comunemente utilizzate sia per la ricerca di base della biologia degli organismi che per la ricerca applicata. L'uso delle tecnologie del DNA per modificare i geni per scopi pratici è detta ingegneria genetica. In particolare, l'ingegneria genetica trova larga applicazione nell'area delle biotecnologie, settore che comprende qualsiasi tecnica, applicata a sistemi biologici o a organismi viventi, per realizzare o modificare prodotti o processi per uno specifico scopo. Difatti le biotecnologie comprendono manipolazioni che non implicano necessariamente le tecnologie del DNA ricombinante, come ad esempio l'uso del lievito per produrre birra e fare il pane, e l'uso di batteri per fare lo yogurt e formaggio.
Clonaggio del DNA
Un clone è una linea di cellule o individui geneticamente identici, derivati da uno stesso progenitore.
Durante il processo di mitosi si generano due cellule geneticamente identiche tra di loro e al progenitore. La mitosi è un processo naturale che porta alla formazione di un clone. Quando un batterio duplica segue lo stesso principio, genera cellule geneticamente identiche tra di loro (colonia batterica).
Il clonaggio del DNA è un metodo per produrre molte copie di un frammento di DNA, quale ad esempio un gene d'interesse. Questa tecnica è inoltre utilizzata anche per clonare importanti sequenze di DNA che non codificano per proteine, come ad esempio sequenze regolatrici dell'espressione dei geni, promotori, o altre sequenze regolatrici, come gli enhancer.Procedura del clonaggio
- Isolare il DNA da clonare (ad esempio dalla cellula)
- Tagliare il DNA in frammenti con un enzima di restrizione.
- Aprire il plasmide batterico circolare, nello stesso modo del DNA da clonare, quindi con un enzima di restrizione.
- Unire i frammenti di DNA con i plasmidi aperti per generare molecole di DNA ricombinante (vettore + frammento di DNA), utilizzando la DNA ligasi.
- Introdurre le molecole ricombinanti in cellule batteriche, le quali replicando permettono ai vettori al loro interno di replicare, in modo da amplificare ciascun vettore e quindi ciascun frammento di DNA. Si sfrutta la capacità trasformante dei batteri; le cellule competenti sono le cellule in grado di captare DNA dall'ambiente esterno (l'efficienza trasformante è molto bassa, perché sono poche le cellule batteriche competenti, in grado di captare DNA dall'esterno).
O.I- Vettore Frammento di DNA Molecola di DNA ricombinante Cellula batterica 2 Trasporto al'interno della cellula ospite Batterio che porta il DNA ricombinante 3 Moltiplicazione della molecola di DNA ricombinante 1 1 4 Divisione della cellula ospite 5 Numerose divisioni cellulari danno origine a un clone Colonie batteriche cresciute su mezzo di coltura solido
- Individuare i batteri contenenti il DNA ricombinante.
- Far replicare le cellule batteriche che hanno internalizzato il DNA ricombinante su un terreno di coltura solido (agarosio). Ciascun batterio trasformato prolifererà e darà luogo ad una colonia di cloni.
Applicazioni del Clonaggio nella Ricerca di Base
- Determinare la sequenza di geni
- Determinare le sequenze regolatrici
- Studiare la funzione dei geni
- Studiare la regolazione dei geni
Studiare il prodotto dei geni
- Manipolare i geni e/o la loro regolazione
Applicazioni del Clonaggio nella Ricerca Applicata
Terapia genica volta a correggere o trattare una malattia genica: se una malattia è causata dal mal funzionamento di un gene, possiamo correggere questo mal funzionamento del gene trasferendo (o sostituendo) nell'organismo affetto il gene che funziona in maniera normale.
- Diagnosi di malattie genetiche: si studia la sequenza di DNA
DNA fingerprinting in medicina legale: test di paternità Produzione di farmaci (Humulin, NGF, plasminogeno ecc)
- Generazione di ceppi batterici, animali e piante geneticamente modificati
Vettori Plasmidici di Clonaggio
I vettori plasmidici di clonaggio sono molecole di DNA di piccole dimensioni, a loro volta ricombinanti perché sono state ingegnerizzate per poter avere determinate caratteristiche da poter essere utilizzate durante il clonaggio. In generale, i vettori di clonaggio sono molecole di DNA ricombinante in grado di replicare in cellule batteriche.
I vettori di clonaggio sono in grado di autoreplicarsi, in questo modo replicano anche il frammento di DNA che hanno internalizzato. I vettori di clonaggio possono essere plasmidi o fagi. I fagi sono dei virus in grado di infettare solo cellule batteriche; alcuni genomi di questi virus possono essere manipolati e utilizzati come vettori.I vettori di clonaggio sono piccole molecole di DNA circolare (alcune sono lineari) a doppio filamento (alcuni possono essere a singolo filamento). I vettori di clonaggio esistono come strutture separate dal cromosoma, che derivavano da plasmidi naturali, che poi sono stati ingegnerizzati (sono state rimosse tutte quelle sequenze non essenziali).
I vettori di clonaggio si distinguono in High copy number (10-100 copie/cellula) e Low copy number (1-4 copie/cellula): una volta all'interno dei batteri, queste molecole possono essere presenti in un numero di copie basso, come 1-4 copie per cellula, o in un numero di copie alto, come 10-100 copie per cellula. I vettori di clonaggio hanno un'elevata capacità trasformante (sono comunque poche le cellule batteriche competenti). (i vettori di clonaggio devono avere una dimensione contenuta).
Caratteristiche dei Vettori di Clonaggio
Tutte le molecole di DNA per poter essere usate come vettori devono avere delle caratteristiche ben precise:
Origine di replicazione -> che permette la replicazione del plasmide stesso
- Siti unici per gli enzimi di restrizione (ripetuti una volta sola)
- Uno o più markers di selezione (geni di resistenza agli antibiotici)
Esempi di Vettori di Clonaggio
Il primo vettore utilizzato è il pBR322. Questa è una molecola di origine plasmidica, quindi ha un DNA circolare a doppio filamento, lungo 4362 bp. Nella sua rappresentazione sono evidenziate le sequenze essenziali:
EcoRI
- Origine di replicazione -> fondamentale affinché un vettore possa essere replicato in una cellula ospite, perché lo scopo del clonaggio è avere una notevole quantità di DNA ricombinato.
Elementi di selezione -> (Amp' e Tet'), che sono geni che conferiscono resistenza agli antibiotici (resistenza alla ampicillina e resistenza alla tetraciclina) in modo da selezionare solo i batteri che hanno acquisito resistenza agli antibiotici.
- Siti unici per gli enzimi di restrizione (PstI, EcoRI, HindIII, BamHI, SalI) -> i quali riconoscono e tagliano il vettore in modo sequenza-specifico. Sono importanti perché per inserire un frammento di DNA all'interno del vettore bisogna tagliare il vettore ma è importante che i siti siano unici in modo da far tagliare l'enzima di restrizione solo in un punto. HindIII BamHI Ampľ Tet PstI pBR322 Sall Origin of replication
Un altro vettore di clonaggio è pUC19, lungo 2.68 Kbp è più piccolo del precedente:
- Origine di replicazione
- Resistenza all'ampicillina (marker di selezione Amp')
- Gene Lacl e gene LacZ' che derivano dall'operone Lac (lacZ' permette di selezione solo i plasmidi che hanno internalizzato il gene di interesse) lacZ' gene Ampr Multiple cloning sequence pUC19 2.68kbp lacl gene Origin of replication
MCS (Multiple cloning sequences o polylinker) -> è una regione molto importante per la strategia di clonaggio. È una regione ingegnerizzata del vettore in cui sono presenti tutti i siti unici per gli enzimi di restrizione; questa ingegnerizzazione permette di semplificare il processo di clonaggio, in modo tale da avere una regione ben precisa in cui inserire i frammenti di DNA.