Editing del genoma: tecniche ZFN, TALEN e CRISPR/Cas9 e applicazioni
Documento sull'editing del genoma, descrivendo le tecniche ZFN, TALEN e CRISPR/Cas9. Il Pdf, adatto per lo studio universitario in Biologia, esplora le applicazioni di queste tecnologie nella terapia genica per l'anemia falciforme, le beta-talassemie e le degenerazioni retiniche, supportato da diagrammi esplicativi.
Mostra di più11 pagine
Visualizza gratis il Pdf completo
Registrati per accedere all’intero documento e trasformarlo con l’AI.
Anteprima
EDITING DEL GENOMA
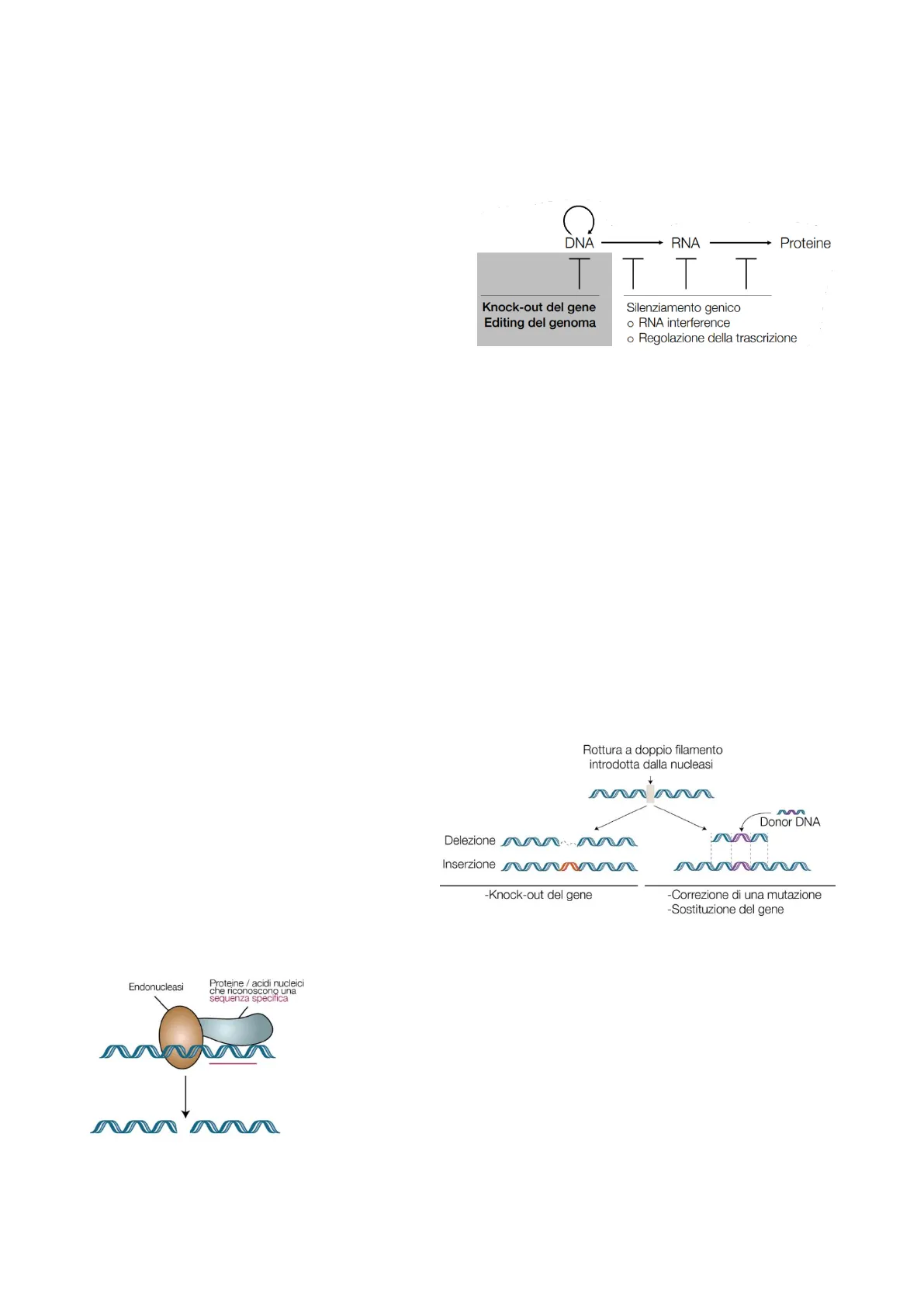
La regolazione dell'espressione genica può avvenire su vari livelli del flusso genico: interferendo con la stabilità dell'mRNA; DNA operando alterazioni nei processi di T trascrizione o traduzione. Ad esempio, Knock-out del gene Editing del genoma attraverso i siRNA si ottiene un silenziamento dell'espressione genica in quanto si va a bloccare I'mRNA all'interno della cellula, downregolando l'espressione. Proteine RNA TTT Silenziamento genico o RNA interference o Regolazione della trascrizione
L'editing del genoma prevede l'intervento al livello del DNA stesso: modifiche operate sul genoma stesso sono modifiche permanenti. Tutte le modifiche invece che avvengono al livello post-trascrizionale sono di carattere temporaneo. Questa tecnica permette di modificare, inserire o cancellare una porzione del DNA in siti specifici del genoma; andando ad alterare in maniera permanente il genotipo e, conseguentemente, il fenotipo dell'organismo.
Tra le varie applicazioni si ha lo studio funzionale di geni; modificazione di geni nell'ambito di agricoltura o correzione di geni coinvolti nelle malattie umane.
Qualsiasi tecnica dell'editing del genoma pone le proprie basi sui meccanismi di riparazione delle rotture a doppio filamento, indicati anche come DSB.
Tra di essi si riconoscono due meccanismi principali:
- Non-homologous end joining (NHEJ): le estremità rotte vengono risaldate senza un DNA stampo omologo; si verifica Rottura a doppio filamento introdotta dalla nucleasi quindi una inserzione/delezione casuale che porta al knock out del gene d'interesse.
- Homology directed repair (HDR): le estremità rotte vengono risaldate Inserzione in presenza di un DNA stampo omologo; si ottiene una correzione o sostituzione con la forma corretta del gene target Delezione Donor DNA -Knock-out del gene -Correzione di una mutazione -Sostituzione del gene
Endonucleasi I processi di riparazione, e quindi di fatto tutti i processi Proteine / acidi nucleici che riconoscono una sequenza specifica d'editing, prevedono la partecipazione di un complesso di endonucleasi, le quali si occupano del taglio al livello della doppia elica, e proteine o acidi nucleici, i quali sono in grado di riconoscere specifiche sequenze al livello del DNA. Le proteine riconoscono la sequenza e, legandovisi, indirizzano nel corretto sito le endonucleasi in quanto sono enzimi poco specifici. Il tool biologico di endonucleasi e proteine permette che si operi un taglio specifico sul genoma.
Si riconoscono 3 principali tecniche di editing genomico: ZFN; TALEN; CRISPR1.ZFN
Tecnica ZFN
È la prima tecnica sviluppata dal punto di vista cronologico.
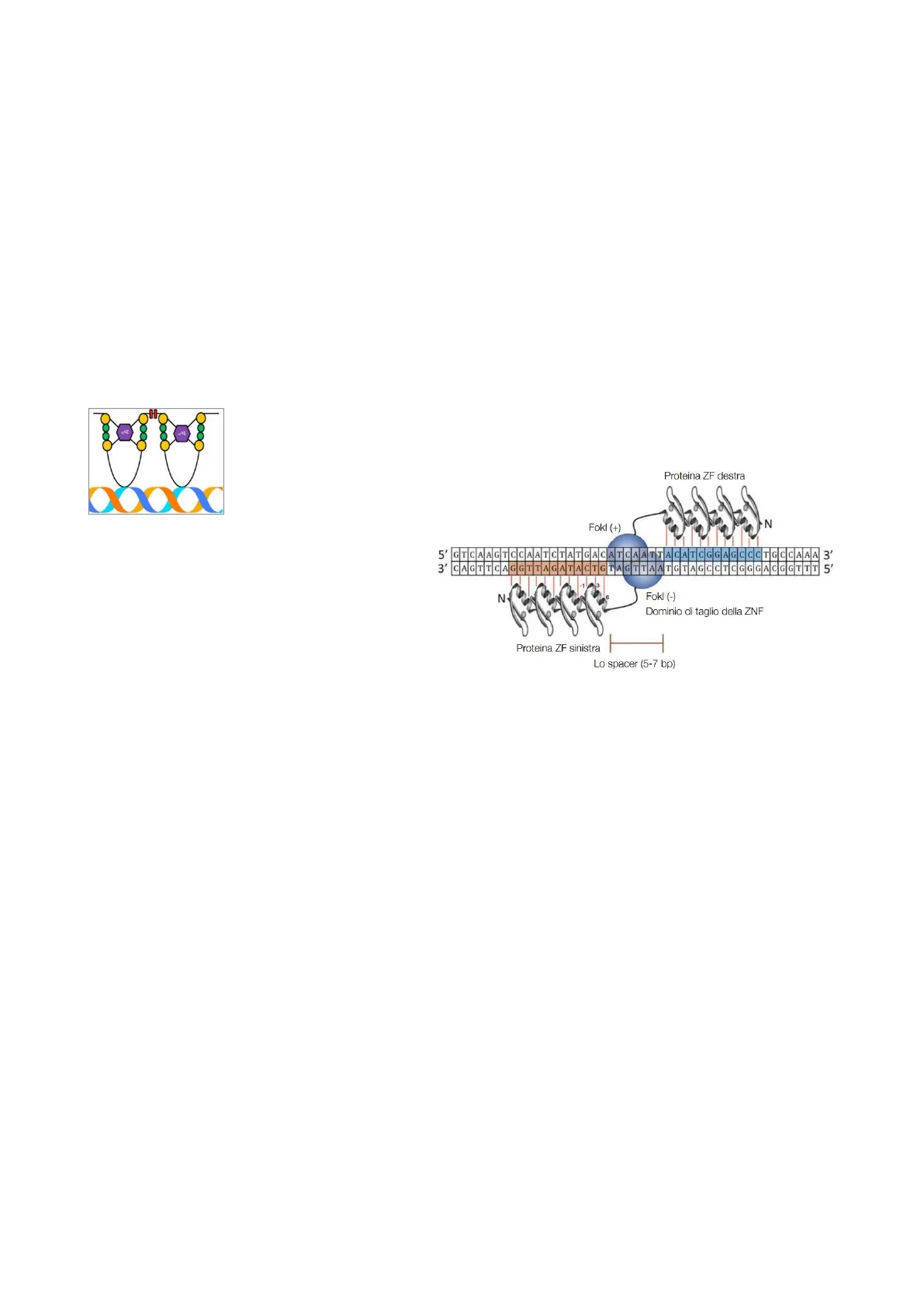
ZFN prevede l'utilizzo dell'endonucleasi di restrizione Fokl, la quale è caratterizzata da avere un dominio N-terminale in grado di legare il DNA e un dominio C-terminale nel quale risiede il sito di taglio aspecifico del DNA stesso.
L'enzima è funzionale in forma dimerica; in particolare, grazie all'N-terminale, Fokl lega il sito di riconoscimento 5'-GGATG-3' sul dsDNA ed opera un taglio aspecifico al livello di sequenza. Si ottiene un taglio sul primo filamento a 9 nucleotidi a valle della sequenza di riconoscimento di 5 nucleotidi e un taglio sul secondo filamento circa 13 nucleotidi a monte rispetto al sito.
Affinché si possa parlare di editing del genoma, il taglio deve esser operato in maniera sequenza specifica. Ciò è ottenuto in quanto il dominio di taglio non- Proteina ZF destra specifico di FokI può essere usato per Fokl (+) N costruire nucleasi ibride aventi siti di legame al DNA più specifici; queste sono 5'GTCAAGTCC ACA 3' CAGTTCAGGTTAGATACTGTAG le nucleasi a dita di zinco, ZFN. La struttura proteica delle dita di Zn guida l'endonucleasi Fokl a tagliare il genoma in maniera sequenza-specifica. CGGAGCCCTGCCAAA 3' ATGTAGCCTCGGGACGGTTT 5' N- Fokl (-) Dominio di taglio della ZNF Proteina ZF sinistra Lo spacer (5-7 bp)
Essendo la FokI un dimero, ciascun monomero risulta esser legato a un motivo dita di Zn. Il motivo di fatto è in grado di riconoscere la sequenza sia sullo strand positivo che su quello negativo del DNA.
Col fine di creare il tool biologico in grado di correggere il genoma, è necessario ingegnerizzare una proteina ZF in grado di riconoscere specifiche sequenze. Tutto ciò prevede molto lavoro.
Tecnica TALEN
La tecnica TALEN è stata sviluppata quasi in parallelo alla tecnica ZFN.
Si basa sull'utilizzo di proteine naturali, le TAL, le quali sono secrete dai batteri Xanthomonas.
Il batterio, attaccando la pianta, inietta le TAL all'interno delle cellule vegetali fino ad arrivare al nucleo, dove attivano la trascrizione di geni specifici che rendono la pianta più suscettibile all'infezione.Il dominio del TAL che lega il DNA è formato da una sequenza altamente conservata di 33-34 aa ripetuti; il 12º e 13º aa sono divergenti, indicati come RVD. Sono di fatto la porzione estremamente variabile, correlata al riconoscimento specifico dei nucleotidi sul DNA bersaglio. Affinché vi sia un riconoscimento specifico della sequenza bersaglio è necessario costruire in vitro dei TALEN con opportune RVD.
Fokl GTTCACTATA ANNANGCGGTAATGAT CAAGTGATANNNN NCGCCATTACT Fokl TALEs bind single nucleotides: = A = T = G = C TALEN sinistra Spacer 16-19 bp TALEN destra
Il funzionamento dei TALEN è molto simile a quello dei ZF. Entrambi i sistemi sfruttano l'idea di base di operare con tools biologici composti da endonucleasi (FokI) guidate a tagliare in maniera specifica da apparati proteici o di acidi nucleici. In seguito al taglio del doppio filamento si verificano i processi di riparazione NHEJ o HDR.
Fokl ZFN TALEN Fokl HDR NHEJ HDR Knock-out del gene Correzione di una mutazione Sostituzione del gene
Queste tecniche di editing richiedono una grande mole di lavoro in quanto vi è la necessità di modificare la sequenza codificante per le proteine ZF che per gli effettori TAL attraverso clonaggio molecolare; successivamente si deve andare a purificare la proteina modificata e, insieme alle endonucleasi, usarla poi nell'editing. Si è cercato quindi di trovare una tecnica che richiedesse minor lavoro; circa 15 anni fa si è arrivati alla realizzazione di CRISPR, tecnica che ha rivoluzionato il mondo della ricerca.
La tecnica TALEN rimane comunque quella con maggior tasso di successo grazie all'alta specificità degli effettori TAL.
Tecnica CRISPR
Il sistema CRISPR si basa su strutture biologicamente naturali, già esistenti nei batteri che servivano come difesa contro le infezioni virali. È una sorta di sistema immunitario del batterio.
Queste sequenze furono scoperte nel 1987 in E. coli; nel 2000 furono identificati simili cluster in altri batteri e archeobatteri; la denominazione CRISPR risale al 2002.
Ripetizione Spacer RipetizioneCRISPR sta ad indicare segmenti di DNA contenenti brevi sequenze ripetute; ogni ripetizione è seguita da brevi frammenti di DNA spaziatore. Quest'ultimi sono il residuo genomico del virus che precedentemente aveva infettato la cellula batterica; di fatto rappresentano una sorta di memoria d'immunità del batterio riguardo le infezioni virali che riesce a sconfiggere.
Il tool comprende le proteine CAS, ossia proteine associate alle sequenze CRISPR; sono sostanzialmente enzimi quali nucleasi ed elicasi in grado di operare tagli sul DNA.
Tutto il cluster di sequenze CRISPR e proteine CAS forma il sistema CRISPR/CAS.
sgRNA (tracrRNA-crRNA chimera) Genome specific SgRNA sequence Cas Genomic DNA Phase1: immunization Infection PAM(5'-NGG-3") Cas9 Nuclease Viral DNA Nel batterio questo sistema rappresenta una via di difesa: Cleavage Spacer l'infezione virale primaria Insertion of a new repeat- spacer unit produce le sequenze CRISPR; il Cas complex > batterio reagisce alle successive Type II TracrRNA Cas9 Cas1 Csn2 CRISPR locus Cas2 infezioni virali trascrivendo le FATFAT Repeats Spacers sequenze spacer e repeat di DNA adiacenti in una unica lunga Pre-CRISPR trascritto RNA TracrRNA trascritto JUUL molecola di mRNA pre-CRISPR. Questo RNA si associa ad altri trascritti come TracrRNA e Cas9; TracrRNA e mRNA pre-CRISPR rappresentano una via di difesa batterica per infezioni secondarie. Se si verifica una seconda infezione ad opera dello stesso virus, il batterio sarà in grado di difendersi in quanto andrà a digerire il genoma virale grazie a nucleasi e un apparato guida che indirizza specificatamente la nucleasi stessa sul genoma virale. Di fatto le sequenze pre-CRISPR, derivanti dalla trascrizione anche di sequenze spacer, contengono porzioni di genoma virale; sono le guide per le nucleasi Cas.
SISTEMA CRISPR/CAS9 NEI BATTERI
Il funzionamento ottimale del sistema CRISPR/Cas9 si basa sulla presenza e, ancor di più, sul riconoscimento specifico della sequenza bersaglio del Sequenza bersaglio DNA virale. Esso avviene in quanto nel genoma batterico è Sequenza PAM presente la stessa sequenza bersaglio; ciò che permette la discriminazione tra il genoma batterico con quello virale è la sequenza PAM, che segue sempre la sequenza bersaglio solamente nel genoma virale.
La sequenza PAM, detta anche motivo adiacente al protospacer, è una piccola sequenza di circa 2-6 pb; il taglio ad opera delle nucleasi avviene a 3-4 nucleotidi a monte della sequenza stessa.Altra componente fondamentale nel sistema CRISPR/Cas9 è l'RNA guida. Esso include:
- un trascritto spacer crRNA, il quale permetterà poi il riconoscimento della sequenza bersaglio del genoma virale; + crRNA (crispr RNA) tracrRNA (trans-attivatore crRNA) gRNA (RNA guida)
- un trascritto transattivatore, detto anche tracrRNA; questo è sempre uguale nei vari gRNA in quanto ha la funzione di scaffold.
Ciò che cambia da gRNA in gRNA è il crRNA
Di fatto il gRNA rappresenta l'analogo delle proteine ZF e effettori TAL che si hanno nelle altre tecniche usate inizialmente per l'editing del genoma. Infatti, il ruolo del gRNA è quello di guidare la nucleasi verso uno specifico sito, evitando tagli casuali.
Le nucleasi usate nel sistema CRISPR/Cas9 sono proprio le proteine Cas9, le quali sono dotate di attività nucleasica.
Riassumendo il sistema CRISPR/Cas9 si basa su tre principali componenti:
- Sequenza bersaglio, seguita necessariamente dalla sequenza PAM;
- gRNA;
- nucleasi Cas9; Sequenza Sequenza PAM bersaglio gRNA (RNA guida) DNA del virus o plasmide Cas9
Nel batterio, il sistema derivante dall'unione di Cas9 con l'gRNA è in grado di riconoscere e legare il genoma virale per andarlo a degradare. Questo sistema si ha solo per infezioni secondarie; è a partire dall'infezione primaria che è permessa di fatto la formazione dell'gRNA.
GENE EDITING
Nell'ambito della ricerca, sfruttando le proprietà del sistema CRISPR/Cas9, è possibile ricreare il sistema in vitro grazie alla generazione di vettori Sequenza Vettore Vettore con il gene Cas9 con il gRNA bersaglio PAM per clonaggio trascriventi per la Cas9, la cui trascrizione porta ad una overespressione della stessa, e vettori per Il gene gene Cas9 da modificare l'gRNA, in cui viene incorporata anche la sequenza gRNA bersaglio del gene da modificare.
Il sistema CRISPR/Cas9 è diventato il fiore all'occhiello tra tutte le tecniche di editing genomico in quanto per andare a modificare/eliminare un gene mutato non si deve far altro che operare un clonaggio genico di una sequenza bersaglio, di circa 20 pb, all'interno del vettore.