I Modelli Animali: genetica medica, sperimentazione preclinica ed epigenetica
Documento di Appunti universitari sui Modelli Animali, la genetica medica e l'epigenetica. Il Pdf esplora la genetica medica, analizzando mutazioni e il loro impatto sul fenotipo, la sperimentazione preclinica e l'epigenetica, utile per lo studio della Biologia a livello universitario.
Mostra di più14 pagine

Visualizza gratis il Pdf completo
Registrati per accedere all’intero documento e trasformarlo con l’AI.
Anteprima
La genetica medica e le mutazioni
La genetica medica si studia principalmente perché esistono malattie genetiche causate da mutazioni all'interno del DNA. Queste mutazioni possono avere varie origini e nature, colpire regioni codificanti e non codificanti e avere effetti diversi a seconda del tipo di mutazione. Un tipo di mutazione molto rilevante sono, ad esempio, le mutazioni non-senso e le mutazioni frameshift, che hanno un impatto molto forte sul prodotto genetico e, di conseguenza, un'alta probabilità di avere un forte impatto sul fenotipo. Analizziamo uno studio in cui, grazie alla Next Generation Sequencing, ovvero alle nuove tecnologie che permettono di sequenziare genomi, stiamo facendo grossi passi avanti nella comprensione del genoma, non solo nei pazienti, ma anche in tutti gli individui. In questo studio sono stati sequenziati circa 60.000 soggetti sani, senza malattie evidenti. Quello che è stato trovato è che circa 3.000 di queste persone presentano mutazioni o delezioni, ovvero aberrazioni genetiche. In circa 3.000 persone, sono state riscontrate mutazioni che portano alla perdita del prodotto genico, ma senza alcun fenotipo apparente. In altre parole, il fatto che ci sia una mutazione in un paziente non implica automaticamente che essa causi un determinato fenotipo. Infatti, se sequenziamo il genoma di un paziente con un fenotipo sconosciuto e riscontriamo una mutazione, non possiamo essere sicuri che quella mutazione sia la causa del fenomeno osservato. In realtà, troveremo molte mutazioni che, sebbene possano sembrare rilevanti e potenzialmente causative, potrebbero non essere effettivamente responsabili del fenotipo osservato.
ARTICLE OPEN doi: 10.1038/nature19057 Analysis of protein-coding genetic variation in 60,706 humans Monkol Lek1,2,3,4, Konrad J. Karczewski1.2%, Eric V. Minikel1.2,5%, Kaitlin E. Samocha12.564, Eric Banks2, Timothy Fennell". Anne H. O'Donnell-Luria1-27, James S. Ware2,8,9,10,11, Andrew J. Hill',212, Beryl B. Cummings1,2,5, Taru Tukiainen1,2, Daniel P. Birnbaum2, Jack A. Kosmicki1,2,6.13, Laramie E. Duncan126, Karol Estrada12, Fengmei Zhao !- 2, James Zou?, Emma Pierce Hoffman1.7, Joanne Berghout14,15,David N. Cooper1", Nicole Deflaux17, Mark DePristo18, Ron Do19,20.21,22 Jason Flannick421, Menachem Fromer 6.9.20,24, Laura Gauthier15, Jackie Goldstein1.26, Namrata Gupta7, Daniel Howrigan1,26, Adam Kiezun15, Mitja I. Kurki2.2), Ami Levy Moonshine"", Pradeep Natarajan2-26,27.25, Lorena Orozco2", Gina M. Peloso2-27.28, Ryan Poplin38, Manuel A. Rivas2, Valentin Ruano-Rubio18, Samuel A. Rose", Douglas M. Ruderfer19,20,24, Khalid Shakir!", Peter D. Stenson16, Christine Stevens?, Brett P. Thomas1,2, Grace Tiao15, Maria T. Tusse-Luna30, Ben Weisburd2, Hong-Hee Won" Dongmei Yu6,25.27,22, David M. Altshuler2-1), Diego Ardissino24, Michael Boehnke15, John Danesh, Stacey Donnelly2, Roberto Elosua17, Jose C. Florez2,26,27, Stacey B. Gabriel-, Gad Getz18,26.38,Stephen J. Glatt39,40,4, Christina M. Hultman12. Sekar Kathiresan2,26,20,25, Markku Laakso, Steven MeCarroll",", Mark 1, McCarthy 4/5,46, Dermot McGovern", Ruth McPherson4", Benjamin M. Neale1,46, Aarno Palotie1,2,44º, Shaun M. Purcel][9,20.24, Danish Saleheen5),12 Jeremiah M. Scharf3.6.25,27,32, Pamela Sklar19,20,24,53.54, Patrick F. Sullivan556, Jaakko Tuomilchto", Ming T. Tsuang"". Hugh C. Watkins14.39, James G. Wilson"0, Mark J. Daly1,2,6, Daniel G. MacArthur1-2 & Exome Aggregation Consortiumt Large-scale reference data sets of human genetic variation are critical for the medical and functional interpretation of DNA sequence changes. Here we describe the aggregation and analysis of high-quality exome (protein-coding region) DNA sequence data for 60,706 individuals of diverse ancestries generated as part of the Exome Aggregation Consortium (ExAC). This catalogue of human genetic diversity contains an average of one variant every eight bases of the exome, and provides direct evidence for the presence of widespread mutational recurrence. We have used this catalogue to calculate objective metrics of pathogenicity for sequence variants, and to identify genes subject to strong selection against various classes of mutation; identifying 3,230 genes with near-complete depletion of predicted protein-truncating variants, with 72% of these genes having no currently established human disease phenotype. Finally, we demonstrate that these data can be used for the efficient filtering of candidate disease-causing variants, and for the discovery of human 'knockout' variants in protein-coding genes.
Valutazione della patogenicità delle variazioni del DNA
Per determinare se una variazione nella sequenza del DNA è patogenica, si valutano diversi fattori. Innanzitutto, se una mutazione è stata osservata in pazienti affetti dalla stessa malattia e se un simile fenotipo è stato riscontrato in altri pazienti con lo stesso difetto genetico, è probabile che quella variazione sia la causa della patologia. Un altro indicatore importante è la presenza di mutazioni de novo, cioè varianti genetiche che non sono presenti nei genitori dell'individuo ma che si manifestano nel paziente, suggerendo un'origine spontanea della malattia. Inoltre, se una mutazione è assente in un ampio numero di individui sani, ciò può essere un segnale che essa sia effettivamente legata alla malattia, dato che oggi abbiamo la possibilità di sequenziare il DNA di un numero considerevole di persone sane per fare un confronto. Il tipo di cambiamento genetico è anche un aspetto rilevante, poiché alcune mutazioni, come quelle che alterano la funzione di un gene o la struttura di una proteina, sono più facilmente associate a patologie. Infine, per confermareche una mutazione è causativa di un determinato fenotipo, sono necessari studi funzionali, che permettano di verificare, attraverso esperimenti biologici, l'effetto concreto della mutazione sul funzionamento del gene e sullo sviluppo della malattia.
La Sperimentazione Preclinica
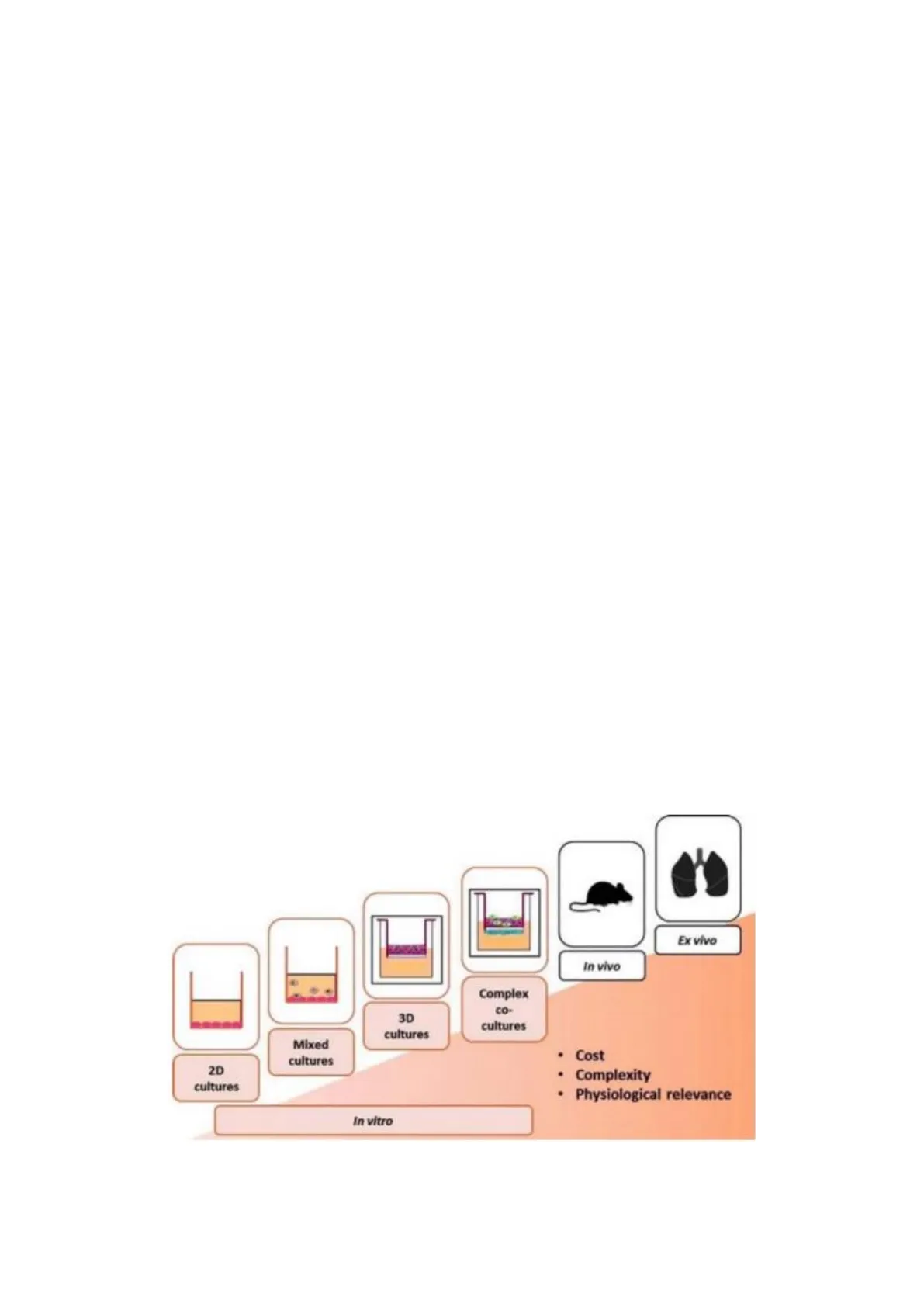
Gli studi funzionali vengono effettuati attraverso quella che viene definita sperimentazione preclinica. Oggi è possibile verificare se una mutazione, introdotta in un animale modello, può riprodurre il fenotipo osservato nei pazienti. Sebbene ci siano mutazioni riscontrate in un alto numero di pazienti con lo stesso fenotipo, che suggeriscono già una relazione causale con la malattia, gli studi preclinici rimangono fondamentali. Se, infatti, in un animale modello viene introdotta una mutazione riscontrata nel paziente e si osserva lo stesso fenotipo, ciò conferma che quella mutazione è effettivamente causativa di quel fenotipo. La sperimentazione preclinica si avvale di modelli che non sono necessariamente animali. In realtà, i modelli sperimentali variano in complessità e rilevanza fisiologica. Il modello animale, complesso, è forse l'ultimo passo della sperimentazione preclinica. All'inizio si utilizzano sistemi più semplici che permettono di acquisire informazioni fondamentali sulla funzione del gene e su come la sua mutazione influenzi la cellula e il sistema cellulare. Ad esempio, si possono condurre studi in vitro per osservare come una mutazione influenzi l'attività enzimatica. In questo caso, si ottiene una proteina ricombinante in vitro e si valuta se la sua attività enzimatica risulta alterata. Un altro esempio è lo studio del legame di una proteina con un recettore o con un ligando.
Successivamente, è possibile passare all'utilizzo di culture cellulari e, in alcuni casi, si possono utilizzare organi isolati da donatori o da animali modello per studiare ciò che accade nell'immunità. Il passo successivo riguarda la sperimentazione sugli animali, attraverso quelli che sono conosciuti come studi in vivo. Con l'aumento della complessità dei modelli, cresce anche la rilevanza fisiologica e patologica dei risultati ottenuti, poiché, ovviamente, studiare una singola cellula non è la stessa cosa che studiare un organismo completo. Tuttavia, questo comporta anche aumenti dei costi, sia in termini economici che di tempo, oltre a porre questioni etiche. Lo studio sugli animali modello è necessario quando non si possono ottenere risposte adeguate in sistemi più semplici; se, però, è possibile evitarlo, è preferibile farlo.
Ex vivo In vivo Complex CO- cultures 3D cultures Mixed cultures • Cost • Complexity 2D cultures • Physiological relevance In vitro
Introduzione Ai Modelli Cellulari In Vitro
La sperimentazione in vitro presenta numerosi vantaggi: è rapida, ha costi relativamente contenuti, permette di coltivare cellule di vari tipi, comprese quelle isolate da pazienti o geneticamente modificate per riprodurre le mutazioni del paziente stesso. In effetti, oggi sono disponibili tecnologie che consentono di modificare il DNA in modo preciso, riproducendo fedelmente la situazione genetica del paziente all'interno di una cellula. Le analisi in vitro sono quindi veloci, i costi sono bassi e non ci sono problematiche etiche, poiché non si utilizzano animali. Inoltre, la semplicità del modello in vitro permette di condurre analisi di segnalazione molecolare o di attività metabolica in modo rapido e meno complesso rispetto a quanto sarebbe necessario per un organismo in vivo. Tuttavia, questa semplicità comporta anche una minor rilevanza dei risultati, che devono comunque essere validati attraverso studi in vivo per ottenere una comprensione più completa della malattia e dei suoi meccanismi.
Modelli cellulari 2D
I modelli cellulari 2D sono tra i più semplici, in quanto consistono in un monolayer di cellule cresciuto su una piastra in un incubatore che mantiene condizioni controllate, come temperatura e concentrazione di CO2. Un tempo, venivano utilizzati fibroblasti prelevati da pazienti o linfoblasti provenienti dal sangue dei pazienti, ma queste colture sono eterogenee e la variabilità tra individui può essere elevata. Oggi si preferiscono cellule immortalizzate, che possono essere di origine tumorale. Sebbene queste cellule siano distanti dal modello fisiologico del paziente, il loro vantaggio è che sono più controllabili, permettendo di introdurre mutazioni e studiarle in un sistema stabile.
Un aspetto importante degli studi in modelli 2D riguarda la trasduzione del segnale, specialmente in malattie metaboliche. In laboratorio, si esamina come la presenza o l'assenza di nutrienti influenzi le cascata di segnali che regolano l'adattamento della cellula e dell'organismo a condizioni metaboliche. Queste risposte vengono alterate dalle mutazioni in geni che codificano per proteine coinvolte in questi percorsi di segnalazione, ed è importante capire il ruolo di questi geni per comprendere lo sviluppo delle malattie metaboliche.
Un altro utilizzo cruciale dei modelli 2D è lo screening farmacologico. Oggi, grazie alla tecnologia, è possibile testare migliaia di farmaci per determinare quale possa correggere un fenotipo patologico. In uno scenario metabolico, ad esempio, un farmaco può ripristinare una situazione che non funziona correttamente in un contesto patologico. Con l'introduzione dell'intelligenza artificiale, è possibile compiere screening in silico, ovvero simulare, tramite modelli computazionali, l'efficacia dei farmaci potenziali. Questo consente di esaminare miliardi di composti per identificare i migliori candidati da testare.
I modelli cellulari 2D possono evolvere in sistemi più sofisticati, come le iPSCs (induced pluripotent stem cells). Queste cellule staminali pluripotenti vengono ottenute dedifferenziando fibroblasti prelevati dal paziente, che tornano a uno stato simile a quello delle cellule staminali pluripotenti. Queste iPSCs possono poi essere differenziate in cellule specializzate, come i neuroni, consentendo lo studio di malattie neurologiche direttamente nel contesto genetico del paziente, senza dover prelevare direttamente neuroni dal paziente.
Modelli cellulari 3D e organoidi
I modelli cellulari 3D, o organoidi, offrono un livello superiore di complessità. Queste colture tridimensionali sono create utilizzando cellule staminali che, in un ambiente di coltura speciale con supporti come la matrice extracellulare, crescono in 3D e si differenziano in cellule specifiche di un determinato organo, come il fegato. Con gli organoidi, è possibile creare modelli più complessi e realistici senza fare ricorso ad animali modello, studiando per esempio il differenziamento neurale in laboratorio.