Diversidad Procariota: Dominio Bacteria, Presentación de Biología
Diapositivas de Universidad sobre Diversidad Procariota: Dominio Bacteria. El Pdf, un recurso de Biología para el grado universitario, explora el árbol filogenético de la vida, las características generales de las bacterias y su morfología celular, incluyendo formas básicas y agrupaciones.
Ver más39 páginas

Visualiza gratis el PDF completo
Regístrate para acceder al documento completo y transformarlo con la IA.
Vista previa
DIVERSIDAD PROCARIOTA
Dominio Bacteria
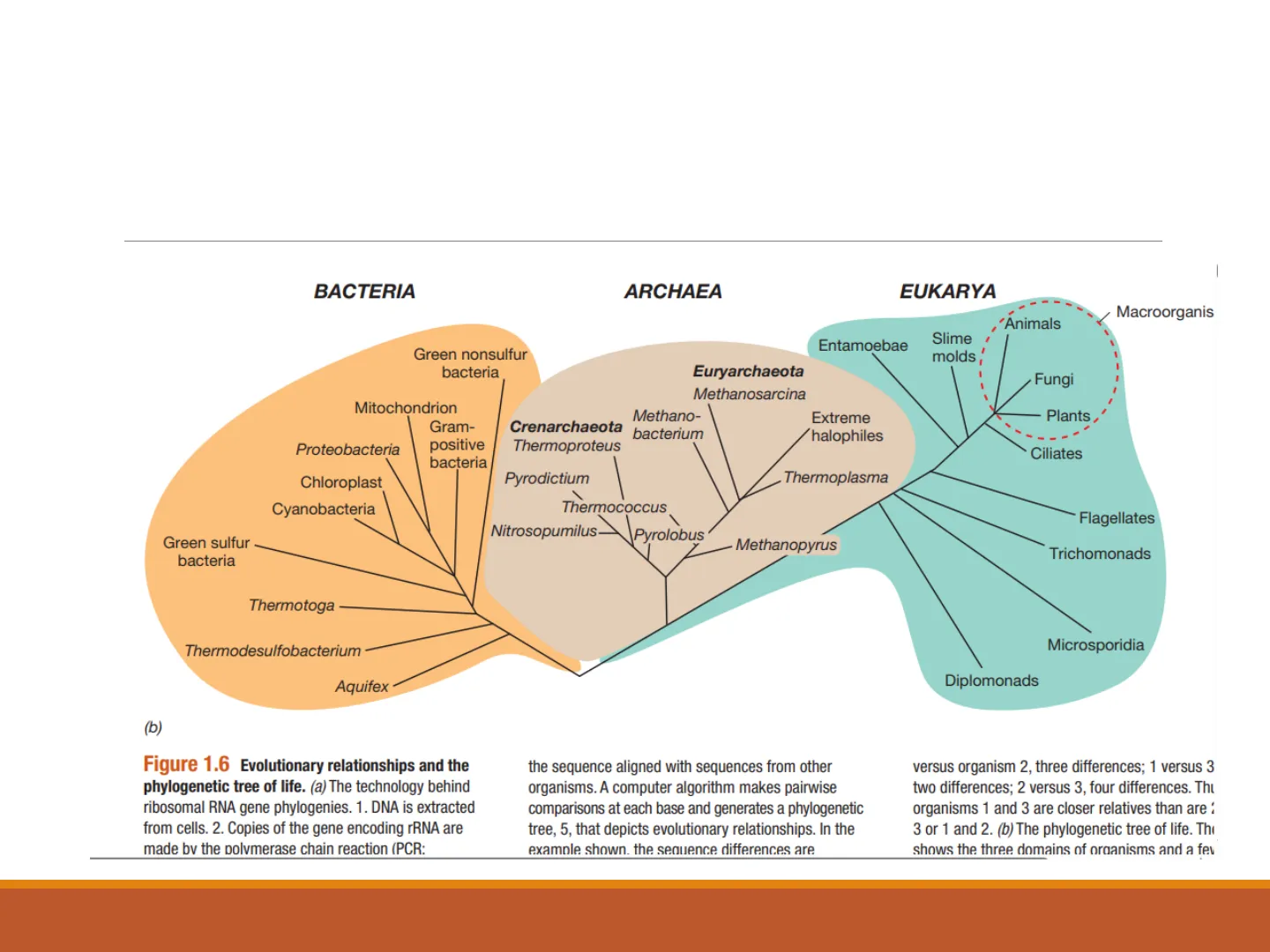
Neyla Benítez Campo Ph.D.Árbol filogenético de la vida
BACTERIA ARCHAEA EUKARYA
Macroorganis 'Animals Entamoebae Slime molds Euryarchaeota Methanosarcina Mitochondrion Methano- bacterium Extreme halophiles Ciliates Pyrodictium Thermoplasma Chloroplast Cyanobacteria Thermococcus Flagellates Nitrosopumilus Pyrolobus Methanopyrus Trichomonads Thermotoga Thermodesulfobacterium Microsporidia Aquifex Diplomonads (b)
Figure 1.6 Evolutionary relationships and the phylogenetic tree of life. (a) The technology behind ribosomal RNA gene phylogenies. 1. DNA is extracted from cells. 2. Copies of the gene encoding rRNA are made by the polymerase chain reaction (PCR: the sequence aligned with sequences from other organisms. A computer algorithm makes pairwise comparisons at each base and generates a phylogenetic tree, 5, that depicts evolutionary relationships. In the example shown. the sequence differences are versus organism 2, three differences; 1 versus 3 two differences; 2 versus 3, four differences. Thi organisms 1 and 3 are closer relatives than are : 3 or 1 and 2. (b) The phylogenetic tree of life. The shows the three domains of organisms and a fev Green nonsulfur bacteria Fungi Plants Proteobacteria Gram- positive bacteria Crenarchaeota Thermoproteus Green sulfur bacteria
BACTERIAS
Organismos procariotas unicelulares, de tamaño muy pequeño: entre 0.5 - 4.0 micras de ancho y menos de 15 micras de largo.
EF6691 5.0 KV X15. 0K 2.000 Magn 13184x 8624.0
Excepciones: Nanobacterias: 0.05-0.2 um Thiomargarita magnifica: 1-2 cm
Morfología celular bacteriana
Coccus Spirochete Rod Hypha Stalk Budding and appendaged bacteria Spirillum Filamentous
Formas Básicas y sus agrupaciones
Cocos
- Coco
- Diplococo
- Diplococo encapsulado
- Pneumococo
- Estafilococo
- Estreptococo
- Sarcina
- Tétrada
Bacilos
- Empalizada
- Cocobacilo
- Bacilo
- Diplobacilo
- Estreptobacilo
Otros Apéndices bacterianos
- Tallo
- Hifa
- Bastón Corynebacteriaceae
- Hélice Helicobacter pylori
- Sacacorchos Borrelia burgdorferi
- Filamento
- Espiroqueta
- Barra alargada Fusobacterium
- Vibrio
- Coma Bdellovibrio
PLEOMORFISMO
La forma de las bacterias varía dependiendo de los nutrientes del medio
TAMAÑO RELATIVO
Figure 3.2 Relative sizes 0 Atom Amino acid Diameter of DNA molecule Protein Ribosome Virus Chlamydia, Rickettsia Mitochondrion Chloroplast Epithelial cell Large protozoan Human egg Human heart Dog (German shepherd) Yeast - - YYYW 0.1 nm 1 nm 10 nm 100 nm 1 pm 10 pm 100 µ.m 1 mm 10 mm 100 mm 1 m 10 m Transmission electron microscope Compound light microscope Unaided human eye Scanning electron microscope 12 XXXX 0 Bacterium Red blood cell, Cell nucleus Tick
ESTRUCTURAS INTERNAS
Citoplasma: Limitado por la membrana citoplasmática, en él se encuentra el nucleoide, los ribosomas y las inclusiones.
Nucleoide: Compuesto por dos cadenas de ADN, formando el cromosoma bacteriano. Generalmente es circular.
Ribosomas: Son las estructuras celulares donde se sintetizan las proteínas. (70S). Posee proteínas y ARNr.
Chromosome Pilus (fimbria) Ribosomes Inclusion Flagellum Capsule or slime layer Plasmid Cell wall Cytoplasm Cell membrane
Plásmido: DNA extracromosómico pequeño, generalmente circular, codifica por propiedades metabólicas especiales.
Endosporas: Estructuras de resistencia de algunas especies de la división Firmicutes (Bacillus y Clostridium).
Chromosome Pilus (fimbria) Ribosomes Inclusion Flagellum Capsule or slime layer Plasmid Cell wall Cytoplasm Cell membrane
Endosporas
Endospore (a)
Presente en algunas bacterias Gram positivas Ej. Bacillus y Clostridium
Proceso de esporulación
- Es un mecanismo de sobrevivencia o protección a: desecación, radiación, desinfectantes, al calor, a la lisozima, a escasez de nutrientes.
- Bajo condiciones favorales ocurre el proceso de germinación: Activación - Germinación - Desarrollo célula vegetativa
Ciclo vegetativo y de esporulación
Peptidoglycan fragments Favorable conditions Germination Free spore Spore coat Cortex Lysis of mother cell VEGETATIVE CYCLE SPORULATION CYCLE Spore coat Axial nucleoid Core Spore
Proceso de formación de endosporas
- Nucleodie alargado axial
- Los dos cromosomas se separan
- Formación del core (Acido dipicolínico, Ca++ y proteínas)
- Septo de la endospora
- Formación del cortex: Síntesis de peptidoglucano
- Formación de cubierta de la endospora
- Formación de exosporium (proteínas-lípidos)
Endospore septum Binary fission Double membrane Cell membrane Spore membrane
Endosporas teñidas con verde de malaquita
1 Célula vegetativa Endospora 10um
Inclusiones
Forman gránulos y otras inclusiones. Almacenan polifosfato, azufre y carbonato Almacenan energía o precursores estructurales de macromoléculas. Ej. Poli-ß-hidoxialcanoatos (PHA) Poli-3-hidoxibutirato
ESTRUCTURAS EXTERNAS
CH3 CH2 H&CN-CH3 CH2 O T 1 O- P=0 Hydrophilic head CH2-CH-CH2 -0-0- O 1 o=c o=c 1 CH2 CH2 - CH2 1 CH2 CH2 CH2 CH2 CH2 CH CH2 1 Hydrophobic tails CH CH2 1 CH2 CH2 1 CH2 CH2 CH2 CH2 CH2 1 CH2 1 CH3 CH3
MEMBRANA CELULAR
Bicapa de fosfolípidos y proteínas. Función: a) Barrera osmótica de la célula. b) Centro de conservación de energía en procariotas c) Anclaje de proteínas
(a) Extracellular space Glycoproteins Glycolipid Lipid bilayer membrane Cholesterol Protein Cytoplasm Phospholipid molecule Hydrophilic ends Hydrophobic ends (b)
PROTEÍNAS TRANSPORTADORAS DE MEMBRANA
Out In Uniport Antiport Symport
Figure 2.21 Structure of membrane-spanning transporters and types of transport events. Membrane-spanning transporters are made of 12 @-helices (each shown as a cylinder) that aggregate to form a channel through the membrane. Shown are examples of three different transport events: uniport, antiport, and symport. Red discs represent the transported molecule; yellow discs represent the cotransported molecule.
Mesosoma
Invaginación de la membrana celular donde se realizan los procesos de respiración y fotosíntesis en procariotas. Participa en la formación del tabique en la división celular y funciones secretoras
Tabique transversal Membrana citoplasmática Pared celular Citoplasma Región nuclear (nucleoide) Ribosomas Mesosomas
PARED CELULAR
Capa rígida que envuelve la célula bacteriana, se encuentra por fuera de la membrana celular. Función: Estructura de protección contra lisis osmótica, responsable de la rigidez y forma celular. Protección contra patógenos y toxinas. Puede contribuir a su patogenidad Composición: En células del dominio Bacteria esta constituída por Peptidoglucano (mureina) N-acetilglucosamina y Acido N-acetilmurámico. Contiene aminoácidos: L-alanina, D-alanina, D-glutámico y Lisina o Ácido Diaminopimélico (DAP)
Peptidoglucano
N- Acetil murámico N- Acetil glucosamina CH, OH CH. OH 2 - 0 -0 H (B1,4) H H H O H H V H H NH-CEO H NH-C=0 I I CH 3 I CH 3 H3 C- CH I C=o 1 L-Alanina I D-Ácido glutámico I aa 3 1 ad 3 - Ácido meso - diaminopimélico en G(-) L-Lisina en G(+) D-Alanina OH H / O -7 0
Peptidoglucano en bacterias Gram positivas y Gram negativas
L-alanine D-glutamic acid Meso-diaminopimelic acid D-alanine D-alanine G G G G M M M M M M G G M M M G M M M M G M Lipoprotein M M M G G E.coli peptidoglycan M M M L-alanine G G D-glutamine L-Lysine M G M M G Penta glycine bridge G M M G M G M M G G Staphylococcus aureus peptidoglycan M M M G D-alanine D-alanine M
Bacterias Gram positivas
El peptidoglucano es muy grueso, corresponde al 90% de la pared celular. Contiene ácido teicoico (polímeros de glicerol o ribitol) Reciben el nombre de Protoplasto cuando se digiere su pared celular. En el proceso de coloración de Gram toman el color violeta del cristal violeta
Pared celular en bacterias Gram positivas
Wall-associated protein Teichoic acid Peptidoglycan Lipoteichoic acid Cytoplasmic membrane (c)
Figure 2.27 Structure of the gram-positive bacterial cell wall. (a) Schematic of a gram-positive rod showing the internal architecture of the peptidoglycan "cables." (b) Structure of a ribitol teichoic acid. The teichoic acid is a polymer of the repeating ribitol unit shown here. (c) Summary diagram of the gram-positive bacterial cell wall.
Bacterias Gram negativas
- El peptidoglucano es delgado, corresponde al 5-10% de la pared celular
- Posee proteínas
- Poseen Periplasma: Espacio entre la membrana citoplasmática y la membrana externa. periplasmicas muy activas
- Poseen una membrana externa adicional, denominada lipopolisacárido (LPS). Contribuye a la adhesion bacteriana. Restringe la entrada de antibióticos y toxinas. Protección contra las defensas del huesped. Actúa como endotoxina.
- Reciben el nombre de Esferoplasto cuando se digiere su pared celular.
- En el proceso de coloración de Gram toman el color rosado de la safranina
Pared celular en bacterias Gram negativas
O-specific polysaccharide Core polysaccharide Lipid A Protein Out Lipopolysaccharide (LPS) Porin 8 nm Phospholipid Peptidoglycan Lipoprotein Cytoplasmic membrane In (a) Outer membrane Periplasm Cytoplasmic membrane Terry Beveridge Georg E. Schutz Outer membrane Cell wall Periplasm
Comparando la pared de Gram positivos y Gram negativos
uram-posluve uram-neyauve Outer membrane Peptidoglycan Cytoplasmic membrane Protein Protein Leon J. Lebeau (a) (b) Peptidoglycan Cytoplasmic membrane Cytoplasmic membrane Peptidoglycan Outer membrane (c) (d) AUmeda and K. Amako A.Umeda and K. Amako (e)
Coloración de Gram
- Preparing a smear Spread culture in thin film over slide Dry in air
- Heat fixing and staining Pass slide through flame to heat fix Flood slide with stain; rinse and dry
- Microscopy 100x Slide Oil Place drop of oil on slide; examine with 100x objective lens
Procedure 1. Flood the heat-fixed smear with crystal violet for 1 min Result All cells purple 2. Add iodine solution for 1 min All cells remain purple 3. Decolorize with alcohol briefly - about 20 sec Gram-positive cells are purple; gram-negative cells are colorless 4. Counterstain with safranin for 1-2 min G- G+ Gram-positive (G+) cells are purple; gram-negative (G) cells are pink to red (a)