Acidi nucleici: composizione, struttura e sequenziamento Sanger del DNA
Documento di Università sugli acidi nucleici, inclusa la loro composizione e struttura. Il Pdf descrive i nucleotidi, le basi azotate e il legame N-glicosidico, con una sezione dedicata al sequenziamento Sanger e alla struttura secondaria del DNA, utile per lo studio della Biologia.
Mostra di più13 pagine
Visualizza gratis il Pdf completo
Registrati per accedere all’intero documento e trasformarlo con l’AI.
Anteprima
Acidi nucleici
Inizialmente vennero fatte solo delle osservazioni, successivamente vennero fatti anche esperimenti. Uno dei primi fu Mendel, che scoprì il sistema di segregazione dei caratteri in modo molto organizzato. Gli esperimenti più importanti che valutiamo sono quello di Avery, Griffith, Hershey e Chase i quali hanno permesso di comprendere successivamente la struttura del DNA grazie alla cristallografia a raggi X per via dell'intuizione di Watson e Crick. Ci sono poi le regole di Chargaff.
Nucleotidi
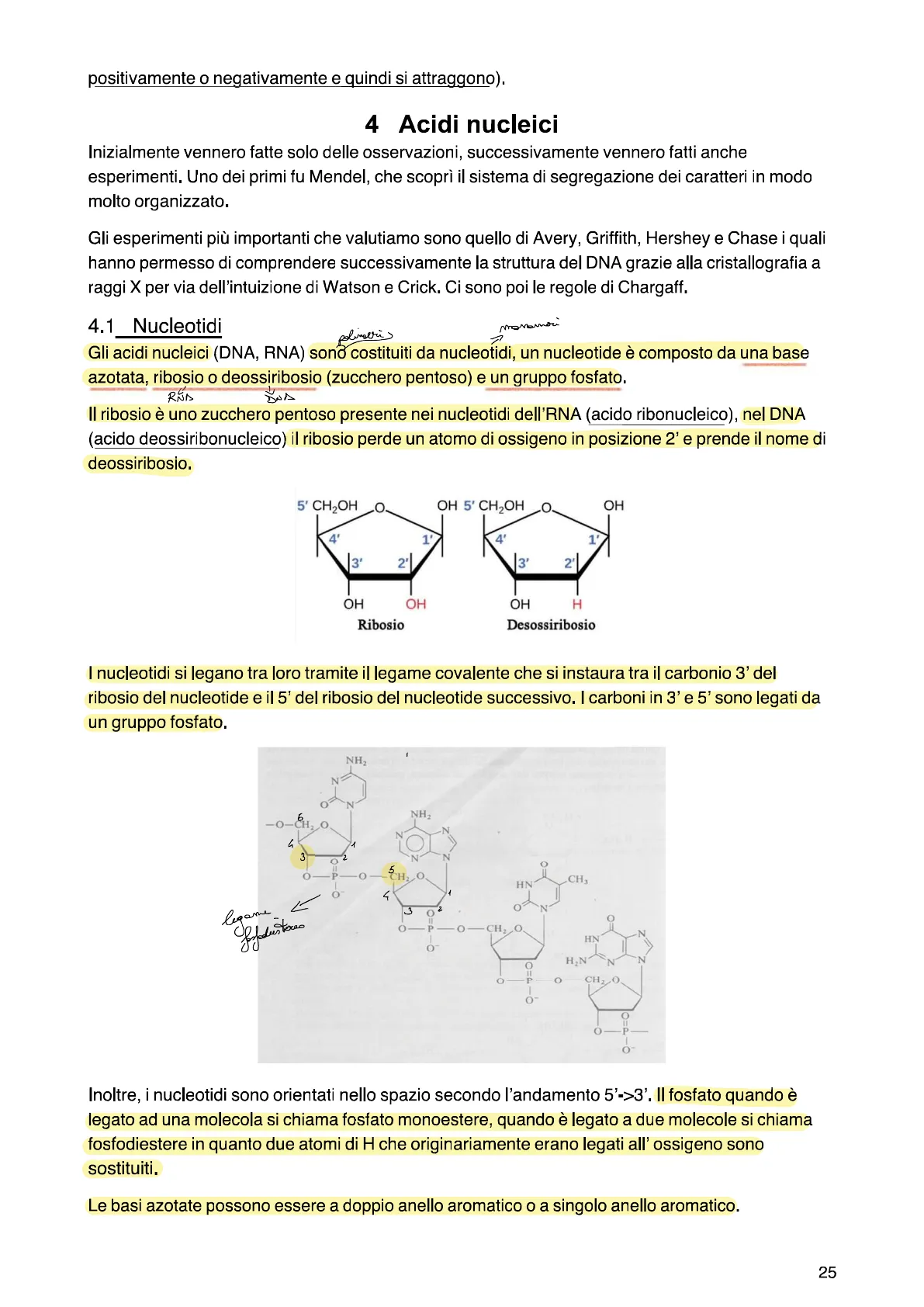
Gli acidi nucleici (DNA, RNA) sono costituiti da nucleotidi, un nucleotide è composto da una base azotata, ribosio o deossiribosio (zucchero pentoso) e un gruppo fosfato. Il ribosio è uno zucchero pentoso presente nei nucleotidi dell'RNA (acido ribonucleico), nel DNA (acido deossiribonucleico) il ribosio perde un atomo di ossigeno in posizione 2' e prende il nome di deossiribosio.
5' CH2OH OH 5' CH2OH .O. OH 4' 1 3' 2'| 3' 2'| OH OH OH H Desossiribosio I nucleotidi si legano tra loro tramite il legame covalente che si instaura tra il carbonio 3' del ribosio del nucleotide e il 5' del ribosio del nucleotide successivo. I carboni in 3' e 5' sono legati da un gruppo fosfato.
1 NH2 N N 6 NH2 -O-CH __ O N N 4 1 3 2 N N O 5 0-P -O CHO CH O 4 1- 0 3 O 0-P-O-CH2_O N HN H,N N O 0-P- O CH __ O -0 O O -P 0 Inoltre, i nucleotidi sono orientati nello spazio secondo l'andamento 5'->3'. Il fosfato quando è legato ad una molecola si chiama fosfato monoestere, quando è legato a due molecole si chiama fosfodiestere in quanto due atomi di H che originariamente erano legati all' ossigeno sono sostituiti. Le basi azotate possono essere a doppio anello aromatico o a singolo anello aromatico.
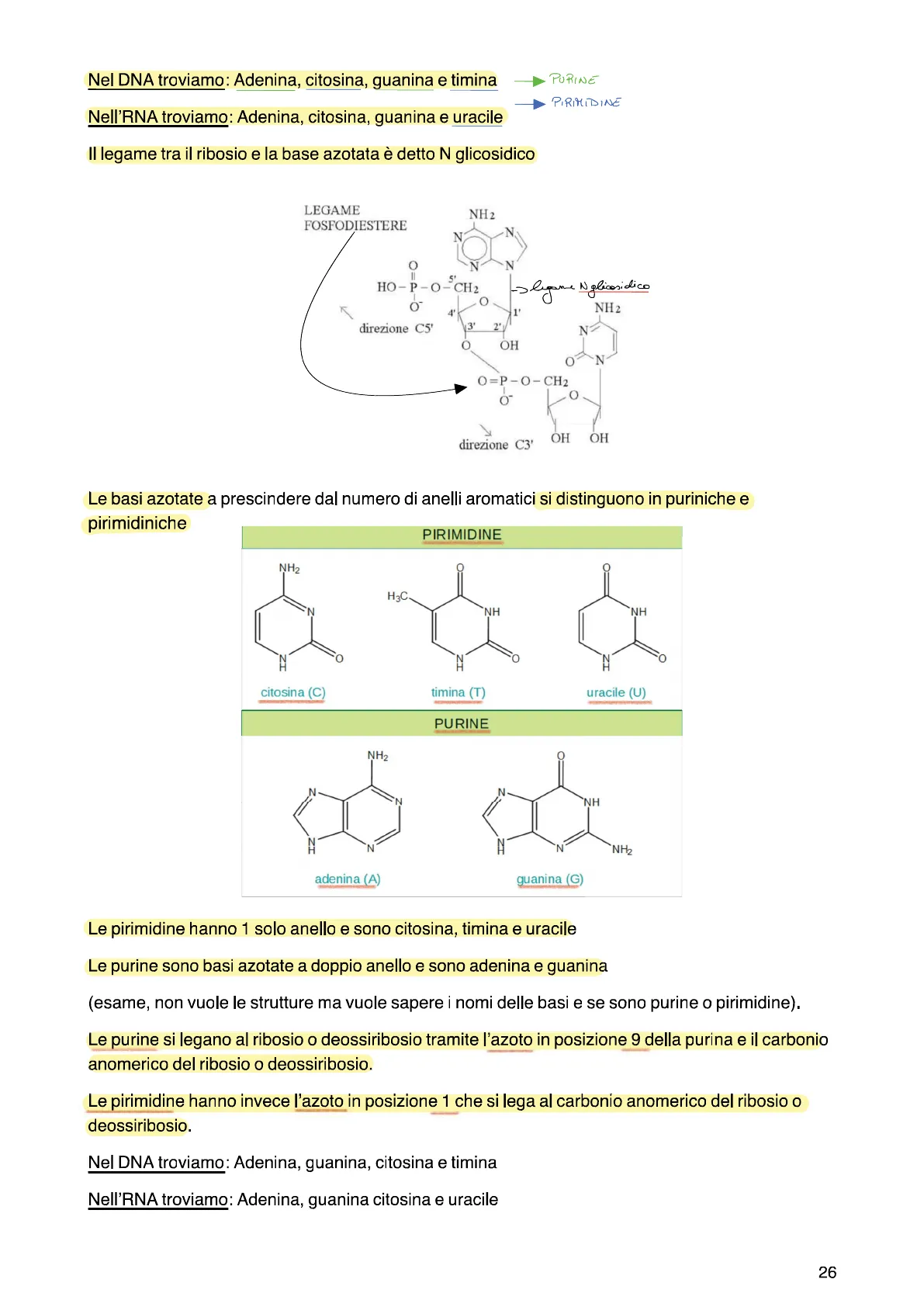
25 legame fosfodustereo 1 2 1 HN 1 Ribosio 4'Nel DNA troviamo: Adenina, citosina, guanina e timina Nell'RNA troviamo: Adenina, citosina, guanina e uracile Il legame tra il ribosio e la base azotata è detto N glicosidico
LEGAME FOSFODIESTERE NH2 N N O N N 5' HO-P-O-CH2 -Dligame Nglicosidico 4 1' 3' 2º N O OH O N O=P-O-CH2 0 -0 OH OH direzione C3' Le basi azotate a prescindere dal numero di anelli aromatici si distinguono in puriniche e pirimidiniche
PIRIMIDINE NH2 H3C N NH NH ZI ZI ZI citosina (C) timina (T) uracile (U) PURINE NH2 N- N NH ZI N N N NH2 adenina (A) guanina (G) Le pirimidine hanno 1 solo anello e sono citosina, timina e uracile Le purine sono basi azotate a doppio anello e sono adenina e guanina (esame, non vuole le strutture ma vuole sapere i nomi delle basi e se sono purine o pirimidine). Le purine si legano al ribosio o deossiribosio tramite l'azoto in posizione 9 della purina e il carbonio anomerico del ribosio o deossiribosio. Le pirimidine hanno invece l'azoto in posizione 1 che si lega al carbonio anomerico del ribosio o deossiribosio. Nel DNA troviamo: Adenina, guanina, citosina e timina Nell'RNA troviamo: Adenina, guanina citosina e uracile
26 O O N NH2 direzione C5'
Nucleosidi
Il nucleoside è caratterizzato dalla base azotata, dal ribosio o deossiribosio e basta. Il nucleotide contiene anche il gruppo fosfato. È importante ricordarlo perché è nomenclatura, quindi se si parla di deossiribonucleosidi abbiamo: deossiadenosina-5-fosfato abbiamo un nucleoside legato al fosfato, abbiamo la deossiguanosina, deossicitidina, deossitimidina. I ribonucleosidi con il ribosio prendono il nome di: adenosina-5-fosfato, guanosina-5-fosfato, citidina-5-fosfato, uridina-5-fosfato
I NUCLEOSIDI = BASE + ZUCCHERO Legame b-glicosidico NH NH N HN HN N H2N HO HO HO HO HO OH HO OH HO OH HO OH adenosine guanosine cytidine uridine NH O CH N HN HN HON" HOS HO HO HO 4º 3 2 HO HO HO HO thymidine 2'-deoxyadenosine 2'-deoxyguanosine 2'-deoxycytidine La rappresentazione della sequenza dei nucleotidi in un polinucleotide è rappresentata come nella foto seguente:
Estremità 5' 5-P O-PO O 5' CH2 0 Base L'orientamento della molecola è sempre 5'->3' La prof all'esame può chiedere il disegno dei nucleotidi.
0- 5' ->5 forfoto Ó-P=0 1 - Q CH2 B 5'CH2 Base H H H 3 H Legame fosfodiesterico O-P=0 1 O 5'CH2 0 Base H H 3 Ò H Ȟ 3-OH Estremità 3' Bisogna quindi mostrare il fosfato in 5', lo zucchero, a quale base è legato, bisogna dire che al 3' è legato un altro gruppo fosfato appartenente al nucleotide che segue. 5' H 3 O H Legame fosfodiesterico O-P-O Ó 5' CH 2 C Base H H H H 3 O I I 3" O-PO Legame fosfodiesterico O H RIBOSIO - -0-P=0 2-0- CH2 3 4 OH 0 1 H 3')-> 3'OH sensibile
27 H H NH2 Ribosio Ribonucleosidi Desossiribosio Deossi-ribonucleosidi H OPer le proteine la struttura primaria è rappresentata dalla sequenza degli amminoacidi legati tra loro da legame peptidico.
Struttura primaria degli acidi nucleici
La struttura primaria degli acidi nucleici è rappresentata dai nucleotidi che si legano covalentemente l'uno all'altro mediante un gruppo fosfato presente tra il 3' del primo nucleotide e il 5' del successivo, inoltre la catena ha l'estremità più alta con il 5' libero (legato a solo 1 gruppo fosfato) e l'estremità più bassa con il 3' libero (legato a un gruppo OH). Nella biochimica, biologia molecolare e bioinformatica le sequenze nucleotidiche vanno sempre lette in direzione 5'->3', inoltre ogni singolo nucleotide viene rappresentato con delle lettere standard a livello internazionale. Per sequenza biologica si intende una sequenza di lettere che rappresenta un polimero nucleotidico o proteico. Se in una posizione c'è l'adenina si usa la lettera A Se in una posizione c'è la guanina si usa la lettera G Se in una posizione c'è la citosina si usa la lettera C Se in una posizione c'è la timina si usa la lettera T Se in una posizione c'è l'uracile si usa la lettera U La sequenza biologica usata per i nucleotidi è rappresentata da almeno 4 lettere che rappresentano le basi azotate che fanno parte del nucleotide, e sono: Nel DNA: A, G, C e T Nell'RNA: A, G, C e U II DNA è composto da un doppio filamento, questi filamenti sono legati tra loro tramite le basi azotate. Ogni base azotata ne ha un'altra complementare:
- Adenina- timina
- A-T
- Guanina- citosina
- G-C
AOG Se voglio intendere che in una posizione c'è una purina si usa la lettera R (alfabeto esteso) SOTTO Se voglio intendere che in una posizione c'è una pirimidina si usa la lettera Y (alfabeto esteso) JAOGOCOTTO Qualsiasi nucleotide si rappresenta con la lettera N oppure X (alfabeto esteso) Se è presente nessuno dei nucleotidi noti si usa il . (punto) (alfabeto esteso) Con l'alfabeto esteso si usa un solo simbolo per sottintendere più nucleotidi che potrebbero essere presenti in un punto. Quindi: Le basi C, T, U si rappresentano con Y Le basi A e G si rappresentano con R A volte si usa anche una lettera come (V, H, D) per indicare più nucleotidi
28L'utilizzo delle lettere Y ed R indica semplicemente la presenza rispettivamente di una pirimidina e una purina, questo può risultare importante in quanto:
- fornisce informazioni sull'ingombro spaziale che è maggiore nel caso delle purine
- Ci permette di capire se la base in questione è complementare ad una purina o una pirimidina, in quanto una purina ha sempre come base complementare una pirimidina e viceversa.
È inoltre importante specificare che la sequenza biologica rappresentata si riferisce agli acidi nucleici, in quanto potrebbe essere anche riferita a una sequenza amminoacidica. Il sequenziamento è la determinazione dell'ordine dei diversi nucleotidi che compongono il DNA o RNA. L'utilizzo dell'alfabeto esteso è più comodo quando si fa il sequenziamento degli acidi nucleici, in quanto durante questa operazione può capitare che non si riesce a risolvere una base e quindi si mette la lettera N, oppure se si capisce che è una purina si mette R, se una pirimidina Y.
Sanger sequencing
Il sequenziamento può essere fatto con varie tecniche, una è il Sanger sequencing, detto anche metodo di terminazione di catena che permette di stabilire la sequenza di nucleotidi nel DNA, con questo metodo viene interrotta la polimerizzazione della catena.
- Quindi inizialmente si usano dei primer specifici per il tratto di genoma da sequenziare
- Poi durante la lettura la DNA polimerasi si bloccherà su ogni nucleotide (se si è fortunati)
- Dopodiché si fa l'elettroforesi per far correre i frammenti di diversa lunghezza sotto un campo elettrico.
Durante l'elettroforesi, in corsie differenti si faceva correre chi si era fermato grazie al D -desossi A, chi si era fermato grazie al D-desossi T, C e G e in base a dove stanno le bande si capisce la sequenza; infatti, si partiva dal basso in cui ci sono i frammenti più corti e via via si risale. Se però la banda non si è formata bene non si capisce se è C o G e per non lasciare il buco viene messo R.
- Se invece si usano i fluorofori il tutto viene messo in un'unica corsia, in quanto la sequenza viene poi letta tramite raggi laser, ogni deossinucleotide avrà poi un colore diverso in base alla base azotata presente grazie al fatto che vengono usati dei marcatori. Anche in questo caso viene fuori un output che dovrà essere letto da un cromatogramma, in particolare un elettroferogramma il quale rileva il deossiribonucleotide tramite la sua assorbanza.
Il ricercatore potrebbe però non essere in grado di risolvere alcune posizioni in modo chiaro. Ogni colore rappresenta una base, per ogni onda ci sono picchi che permettono di ricavare la base, a volte ci sono punti in cui non c'è un picco in nessuna onda in un certo punto e quindi non si sa che base c'è, pertanto si mette la N. Intensità fluorescenza A A AAA T In T -- T - TTT + - T-T-T- G G G G -G - G CN - CT - - - C - - - CC Sequenza CATAGCTGTTT CCTGTGTGAAA Lunghezza oligonucleotide
29Una sequenza ricca di purine ha come controparte una ricca di pirimidine, se c'è un filamento poli- purinico, a volte non conviene scrivere tutte le basi nel dettaglio, ma basta scrivere più volte R in modo da indicare sinteticamente e comodamente la natura delle basi evitando di disperdersi. Anche per gli amminoacidi esiste un alfabeto esteso per indicare gli amminoacidi carichi positivamente, negativamente, polari, apolari, anche in questo caso serve per comprendere comodamente e sinteticamente la struttura. A volte aiuta a comprendere la struttura tridimensionale della stessa. La bioinformatica è stata introdotta nella biologia per sostenere l'analisi sperimentale in modo da creare le strutture 3D o primarie di acidi nucleici e proteine, ma anche per archiviare i dati in banche dati e permettere a tutti di accedervi, risparmiando soldi e tempo. Un ricercatore prima di condividere i suoi dati deve pubblicare un lavoro dimostrando che quel dato è corretto, inoltre chi giudica il lavoro deve dimostrare che il dato sia preciso e che la persona sia conosciuta e abbia esperienza. Oggi una delle analisi più frequenti svolte è quella di usare dati bioinformatici per cercare quali sequenze di dati già esistenti assomiglia a quella nuova del ricercatore. Il formato più usato per rappresentare le sequenze biologiche è il formato FASTA, si usa il nome della sequenza maggiore e si va a capo; è un formato molto semplice che si usa sia per amminoacidi ene acidi nucleici. Oggi però esiste un metodo più sofisticato, ossia le banche dati online Il metodo di confronto tra sequenze è quello di fare la ricerca di una parola in un testo, quindi la biologia ha ereditato dei metodi informatici per fare analisi importanti e via via sempre più spinte, come ad esempio predizioni di strutture, confronti di sequenze, analisi evolutive, capire quanti step evolutivi ci sono voluti per trasformare una sequenza in un'altra.
Struttura secondaria del DNA
La struttura secondaria mette in evidenza che il DNA è fatto da un doppio filamento di polinucleotidi antiparallelo, quindi un filamento avrà il 5' libero in alto e il 3' libero in basso, mentre l'altro filamento ha in alto il 3' libero e in basso il 5' libero.
5 5 3 3 ₸ 0.34nm Y .- (3.4A) 3.4nm (34A) Major groove Minor groove 5' 3' - --- 2nm (20A) 3 5' Le due catene si appaiano tra loro e sono tenute insieme dai legami idrogeno che si formano tra le basi azotate complementari. Esistono delle regole che permettono alle basi azotate di appaiarsi e di rendere i filamenti equidistanti:
- Una purina si appaia sempre con una pirimidina L'appaiamento ideale è tra citosina e guanina (C -G) e tra adenina e timina (A-T), questo perché con queste combinazioni si ottengono il numero massimo di legami idrogeno rendendo la reazione favorita energeticamente rispetto alle altre stabilizzando il DNA. C-G permette 3 legami a idrogeno, A-T permette 2 legami a idrogeno (Scoperta di Watson e Crick).
30